ARN
În chimie, acidul ribonucleic ( ARN în acronim, din engleza RiboNucleic Acid ; mai rar, în italiană , și ARN [1] ) este o moleculă polimerică implicată în diferite roluri biologice de codificare , decodificare , reglare și exprimare a genelor . ARN-ul și ADN-ul sunt acizi nucleici și, împreună cu proteinele , carbohidrații și lipidele , acestea constituie cele patru macromolecule principale esențiale pentru toate formele de viață cunoscute.
La fel ca ADN-ul, ARN-ul este asamblat ca un lanț de nucleotide , dar, spre deosebire de ADN, este mai frecvent în natură ca o singură catenă pliată pe sine, mai degrabă decât o catenă dublă pereche. Organismele celulare folosesc ARN mesager (ARNm) pentru a transmite informații genetice (prin bazele azotate guanină , uracil , adenină și citozină , indicate cu literele G, U, A și C) care direcționează sinteza proteinelor specifice. Mulți viruși își codifică informațiile genetice folosind un genom ARN.
Unele molecule de ARN joacă un rol activ în interiorul celulelor pentru a cataliza reacțiile biologice, pentru a controla expresia genelor sau pentru a percepe și comunica răspunsurile la semnalele celulare. Un astfel de proces activ este sinteza proteinelor , o funcție universală prin care moleculele de ARNm direcționează asamblarea proteinelor în ribozomi . Acest proces folosește moleculele de ARN de transfer (ARNt) pentru a furniza aminoacizii către ribozom, unde ARN ribozomal (ARNr) conectează împreună aminoacizii pentru a forma proteine.
Comparaţie
Structura chimică a ARN-ului este foarte asemănătoare cu cea a ADN-ului , dar diferă în trei aspecte principale:
- Spre deosebire de ADN-ul care este dublu catenar, ARN este o moleculă cu un singur catenar [2] în multe dintre rolurile sale biologice și are un lanț de nucleotide mult mai scurt. [3] Cu toate acestea, ARN-ul poate, datorită asocierii bazelor complementare, să formeze spirale duble intra-elicoidale, ca în cazul ARNt.
- În timp ce ADN conține dezoxiriboză , ARN conține riboză [4] (în dezoxiriboză nu există o grupare hidroxil atașată la inelul pentozei în poziția 2 '). Aceste grupări hidroxil fac ARN mai puțin stabil decât ADN, deoarece este mai predispus la hidroliză .
- Baza ADN complementară a adeninei este timina , în timp ce în ARN este uracil , care este o formă nemetilată de timină. [5]
La fel ca ADN-ul, ARN-ul este biologic mai activ, inclusiv ARNm , ARNt , ARNr , snARN și alți ARN necodificatori , acestea conțin secvențe autocomplementare care permit părților ARN-ului să se plieze [6] și să se conecteze pentru a forma spirale duble. Analiza acestor ANN-uri a arătat că sunt foarte structurate. Spre deosebire de ADN, structurile lor nu sunt formate din spirale duble lungi, ci mai degrabă colecții de spirale asamblate în structuri asemănătoare proteinelor. În acest fel, ARN-ul poate efectua cataliza chimică, ca și enzimele . [7] De exemplu, determinarea structurii ribozomului , o enzimă care catalizează formarea unei legături peptidice , a relevat că situl său activ este compus în întregime din ARN.[8]
Structura

Fiecare nucleotidă de ARN conține un zahăr riboză , cu carbonul numerotat de la 1 'la 5'. O bază este conectată la poziția 1 ', în general, adenină (A), citozină (C), guanină (G) și uracil (U). Adenina și guanina sunt purine , citozina și uracilul sunt pirimidine . O grupare fosfat este atașată la poziția 3 'a unei riboză și la poziția 5' a următoarei. Grupările fosfat sunt încărcate negativ la pH fiziologic, făcând ARN o moleculă încărcată (polianion). Bazele formează legături de hidrogen între citozină și guanină , între adenină și uracil și între guanină și uracil . [9] Cu toate acestea, sunt posibile alte interacțiuni, cum ar fi un grup de baze de adenină legate între ele, [10] sau un tetragiro GNRA care posedă o pereche de baze guanină-adenină. [9]
O caracteristică structurală importantă a ARN-ului care îl deosebește de ADN este prezența unei grupări hidroxil în poziția 2 'a zahărului riboză. Prezența acestui grup funcțional are ca rezultat adoptarea unei spirale în formă de A peste forma B mai frecvent observată în ADN. [11] Acest lucru are ca rezultat un sulcus major foarte adânc și îngust și un sulcus minor superficial și lat. [12] O a doua consecință a prezenței grupării 2'-hidroxil este aceea că în regiunile conformaționale flexibile ale unei molecule de ARN (adică neimplicate în formarea unei duble helice), poate ataca chimic legătura fosfodiester adiacentă și poate cliva coloana vertebrală. [13]
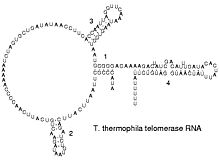
ARN este transcris cu doar patru baze (adenină, citozină, guanină și uracil), [14] dar aceste baze și zaharuri pot fi modificate în numeroase moduri, cum ar fi ARN-urile mature. Pseudouridina (Ψ), în care legătura dintre uracil și riboză se schimbă de la o legătură CN la o legătură CC, și ribotimidina (T) se găsesc în diferite locuri (cele mai importante în ciclul TΨC al ARNt). [15] O altă bază modificată notabilă este hipoxantina, o bază adenină deaminată a cărei nucleozidă se numește inozină (I). Inozina joacă un rol cheie în ipoteza oscilantă a codului genetic. [16]
Există mai mult de 100 de alte nucleozide modificate care apar în mod natural. [17] Cea mai mare diversitate structurală de modificări poate fi găsită în ARNt, [18] în timp ce pseudouridina și nucleozidele deseori prezente în ARNr sunt cele mai frecvente. [19] Rolurile specifice multor dintre aceste modificări ale ARN-ului nu sunt pe deplin înțelese. Cu toate acestea, trebuie remarcat faptul că, în ARN ribozomal, multe dintre modificările post-transcripționale apar în regiuni foarte funcționale, cum ar fi centrul peptidil transferazei și interfața subunității, ceea ce înseamnă că acestea sunt importante pentru funcționarea normală. [20]
Forma funcțională a moleculelor de ARN monocatenar, cum ar fi proteinele, necesită adesea o structură terțiară specifică. Schela acestei structuri este asigurată de elemente structurale secundare care sunt legături de hidrogen în moleculă . Acest lucru duce la mai multe „domenii” recunoscute ale structurii secundare. [21] Deoarece ARN-ul este încărcat, ioni metalici, cum ar fi Mg 2+ , sunt necesari pentru a stabiliza multe structuri secundare și terțiare. [22]
Sinteză
Sinteza ARN este de obicei catalizată de o enzimă, ARN polimeraza , utilizând ADN ca șablon, un proces cunoscut sub numele de transcripție . Transcrierea începe cu legarea enzimei la o secvență promotor din ADN (de obicei „în amonte” a unei gene). ADN-ul dublu catenar este realizat de activitatea helicazei enzimei. Enzima continuă apoi de-a lungul firului șablon în direcția 3 'până la 5', sintetizând o moleculă complementară de ARN în alungirea de 5 'până la 3' care are loc. Secvența ADN determină, de asemenea, locul în care are loc încetarea sintezei ARN. [23]
Prima transcriere a ARN-ului este adesea modificată de enzime după transcriere. De exemplu, o poliadenilare și o acoperire 5 ' sunt adăugate la un pre-ARNm eucariot și intronii sunt îndepărtați din spliceozom .
Există, de asemenea, un număr de ARN polimeraze dependente de ARN care utilizează ARN ca șablon pentru sinteza unei noi catene de ARN. De exemplu, un număr de viruși ARN (cum ar fi poliovirusul ) utilizează acest tip de enzimă pentru a-și replica materialul genetic. [24] Mai mult, ARN-polimeraza ARN dependentă face parte din calea interferenței ARN în multe organisme. [25]
Tipuri de ARN
Există trei tipuri de ARN comune tuturor organismelor celulare :
- ARNm (ARN mesager) care conține informații pentru sinteza proteinelor ;
- ARNr (ARN ribozomal), care intră în structura ribozomilor ;
- ARNt (ARN de transport) necesar pentru traducerea în ribozomi.
În eucariote avem și:
- hnARN (ARN eterogen nuclear) tip de molecule din care face parte pre-ARNm ;
- snARN (ARN nuclear mic) necesar pentru maturarea HnRna.
Sinteza ARN-ului este foarte asemănătoare cu cea a ADN-ului. Cu toate acestea, ARN polimeraza nu necesită un primer. Transcrierea poate începe doar de la o secvență numită promotor și se termină în prezența altor secvențe particulare. S-a sugerat că ARN-ul și-a asumat un rol cheie în organismele primitive înainte de ADN (ARN mondial sau ARN mondial ). În favoarea acestei ipoteze există capacitatea catalitică a unor molecule de ARN ( ribozime ). Informațiile genetice sunt transcrise pe ARNm și apoi utilizate pentru diverse utilizări.
Prezentare generală
ARN-ul mesager (ARNm) este ARN-ul care transportă informații de la ADN la ribozom , locurile de sinteză a proteinelor (traducere) din celulă . Secvența de codificare a ARNm determină aminoacizi secvența proteinei care este produsă. [26] Cu toate acestea, mulți ARN-uri nu posedă cod proteic (aproximativ 97% din producția transcripțională nu codifică proteinele din eucariote [27] [28] [29] [30] ).
Acești așa-numiți ARN necodificatori („ncARN”) pot fi codificați de propriile gene (gene ARN), dar pot fi derivate și din intronii ARNm. [31] Cele mai importante exemple de ARN necodificat sunt ARN de transport (ARNt) și ARN ribozomal (ARNR), ambele fiind implicate în procesul de traducere. [32] Există, de asemenea, ARN-uri necodificatoare implicate în reglarea genelor , procesarea ARN și alte roluri. Unii ARN sunt capabili să catalizeze reacții chimice , cum ar fi tăierea și legarea altor molecule de ARN [32] și cataliza formării legăturii peptidice în ribozom;[8] acestea sunt cunoscute sub numele de ribozime .
În traducere
ARN-ul mesager (ARNm) transportă informațiile unei secvențe proteice către ribozomi, locurile din celula responsabile de sinteza proteinelor . Informațiile sunt codificate astfel încât pentru fiecare trei nucleotide ( codon ) să existe un aminoacid . În celulele eucariote, odată ce precursorul mARN (pre-mARN) a fost transcris din ADN, acesta se transformă pentru a deveni mARN. În această etapă, secțiunile necodificate ale pre-ARNm sunt eliminate. ARNm este apoi mutat din nucleu în citoplasmă , unde ajunge la ribozomi și este apoi tradus în proteina corespunzătoare cu ajutorul ARNt . În celulele procariote, care nu au nucleu și compartimente în citoplasmă, ARNm se poate lega de ribozomi așa cum este transcris din ADN. După o anumită perioadă de timp, mesajul se degradează în componentele sale nucleotidice cu ajutorul ribonucleazei . [26]
ARN de transport (ARNt) este un lanț mic de ARN compus din aproximativ 80 de nucleotide care transferă un aminoacid specific către un lanț polipeptidic în creștere în situsul ribozomal al sintezei proteinelor în timpul traducerii. Posedă situri pentru atașarea aminoacizilor și o regiune anticodonă pentru recunoașterea codonilor care se leagă de o secvență specifică pe lanțul ARN mesager prin legături de hidrogen . [31]
ARN ribozomal (ARNr) este componenta catalitică a ribozomilor. În eucariote, ribozomii conțin patru molecule diferite de ARNr: 18S, 5.8S, 28S și 5S ARNr; trei dintre ele sunt sintetizate în nucleol . În citoplasmă, ARN-ul ribozomal și proteinele se combină pentru a forma o nucleoproteină numită ribozom. Ribozomul leagă ARNm și efectuează sinteza proteinelor. Mai mulți ribozomi pot fi legați la un singur ARNm în același timp. [26] Aproape toți ARN-urile găsite într-o celulă eucariotă tipică sunt ARNr.
ARN-ul mesager de transfer (tmRNA) se găsește în multe bacterii și plastide . Este responsabil pentru marcarea proteinelor codificate de ARNm, cărora le lipsește codonii de stop pentru degradare și previne blocarea ribozomului. [33]
Regulatori de ARN
Diferite tipuri de ARN sunt capabile să regleze în jos expresia genelor pentru a fi complementare unei părți a unui ARNm sau a genei ADN. [34] [35] MicroARN-urile (miARN; 21-22 nt) se găsesc în eucariote și acționează prin interferență ARN ( interferență ARNi sau ARN), unde un complex efector de miARN și enzime capabile să scindeze ARNm complementar blochează traducerea ARNm de la a fi tradus sau accelerează degradarea acestuia. [36] [37]
În timp ce ARN-urile cu interferențe scurte (ARNsi, 20-25 nt) sunt adesea produse în urma descompunerii ARN-ului viral, există și surse endogene de ARNsi. [38] [39] ARNsi acționează prin interferența ARN într-un mod similar cu miARN. Unele miARN și siARN pot provoca metilarea genelor țintă , scăzând sau crescând astfel transcripția acestor gene. [40] [41] [42] Animalele posedă ARN-interacțiune cu piwi (piARN; 29-30 nt) care sunt active în linia germinativă a celulelor și se crede că este o apărare împotriva transpozonilor și joacă un rol în gametogeneză . [43] [44]
Multe procariote au ARN CRISPR , un sistem de reglare similar cu interferența ARN. [45] ARN-urile antisens sunt răspândite, multe reglează o genă , dar unele sunt activatoare de transcripție. [46] O modalitate în care poate funcționa ARN-ul antisens este legarea la un ARNm, formând un ARN dublu catenar care este degradat enzimatic. [47] Există mulți ARN-uri lungi necodificatori care reglează genele din eucariote, [48] unul dintre ele este Xist, care se găsește pe cromozomul X la mamiferele femele și servește la inactivarea acestuia. [49]
In prelucrare
Multe ANN sunt implicate în modificarea altor ANN. Intronii sunt „ biciuiți ” în pre-ARNm de către spliceozomi care conțin mai mulți ARN nucleari mici (snRNA), [5] sau intronii pot fi ribozime care sunt legate prin ele însele. [50] ARN-ul poate fi modificat și prin modificarea nucleotidelor sale cu alte nucleotide A , C , G și U. În eucariote , modificările nucleotidelor ARN sunt de obicei direcționate de ARN nucleolare mici (snoRNA; 60-300 nt), [31] găsite în nucleol și în corpurile Cajal . Enzimele efectuează apoi modificarea nucleotidică. ARNr și ARNt sunt modificate pe larg, dar snARN-urile și ARNm pot fi, de asemenea, ținte pentru modificările de bază. [51] [52] ARN-ul poate fi, de asemenea, demetilat. [53] [54]
Genomul ARN
La fel ca ADN-ul, ARN-ul poate transporta informații genetice. Virușii ARN posedă genomi compuși din ARN care codifică o serie de proteine. Genomul viral este reprodus de unele dintre aceste proteine, în timp ce alte proteine protejează genomul atunci când particula virală se mută într-o nouă celulă gazdă. Viroizii sunt un alt grup de agenți patogeni, dar constau exclusiv din ARN, nu codifică proteine și sunt reproduse de polimeraza unei celule gazdă. [55]
În transcrierea inversă
Virușii cu transcripție inversă sunt capabili să-și reproducă genomul prin transcrierea inversă a copiilor ADN din ARN-ul lor; aceste copii ADN sunt transcrise în ARN nou. Retrotranspozonii s- au răspândit și prin copierea ADN-ului și ARN-ului de la unul la altul [56], iar telomeraza conține un ARN care este folosit ca șablon pentru a face capetele cromozomilor eucariote. [57]
ARN bicatenar
ARN bicatenar (dsARN) este ARN-ul cu două catene complementare, similar ADN-ului care poate fi găsit în toate celulele. ARN dublu catenar (dsRNA), cum ar fi ARN viral sau siARN, poate declanșa interferența ARN în eucariote , precum și răspunsul la interferon la vertebrate . [58] [59] [60] [61] . DsRNA alcătuiește materialul genetic al unor viruși (virusuri ARN dublu catenar).
Anomalii ale ARN-ului
De-a lungul anilor, cercetătorii au descoperit că o mare parte din ARN-ul sintetizat de polimeraze a fost aruncat și doar o mică parte a fost trimisă sub formă de ARNm pentru sinteza proteinelor . De fapt, pentru o proteină medie de aproximativ 400 de aminoacizi (deci 1200 de nucleotide) au fost sintetizate chiar mai mult decât dublul nucleotidelor cu adevărat necesare.
Acest lucru se datorează faptului că în ADN există secvențe care nu se mai codifică, care au fost utilizate de celulă atunci când aceasta nu era încă specializată. Aceste secvențe sunt totuși transcrise de polimerază și se numesc introni , cele care sunt copiate se numesc exoni. Acest fapt implică faptul că, înainte de traducere, acestea vor trebui tăiate; într-un proces numit Splicing
Descoperiri fundamentale în biologia ARN
Cercetările efectuate asupra ARN-ului au condus la numeroase descoperiri biologice importante și au acordat numeroase premii Nobel . Acizii nucleici au fost descoperiți în 1868 de Friedrich Miescher , pe care l-a numit material „nucleino” pentru că i-a găsit în nucleu . [62] Mai târziu s-a descoperit că celulele procariote , care nu au nucleu, le conțin și ele. Rolul ARN-ului în sinteza proteinelor era deja suspectat în 1939. [63] Severo Ochoa în 1959 a câștigat Premiul Nobel pentru medicină (împreună cu Arthur Kornberg ) după ce a descoperit o enzimă capabilă să sintetizeze ARN în laborator. [64] Cu toate acestea, enzima descoperită de Ochoa ( polinucleotid fosforilaza ) s-a dovedit ulterior că este responsabilă pentru degradarea ARN și nu pentru sinteza ARN. În 1956, Alex Rich și David Davies au hibridizat două fire separate de ARN pentru a forma primul ARN a cărui structură ar putea fi evaluată cu cristalografie cu raze X. [65]
Secvența a 77 de nucleotide de ARNt de drojdie a fost descoperită de Robert William Holley în 1965, [66] această descoperire i-a adus premiul Nobel pentru medicină din 1968 (împreună cu Har Gobind Khorana și Marshall Warren Nirenberg ). În 1967, Carl Woese a emis ipoteza că ARN-ul ar putea fi un catalizator și a sugerat că formele de viață timpurii (molecule auto-replicante) ar putea folosi ARN atât pentru a transporta informații genetice, cât și pentru a cataliza reacțiile biochimice: ipoteza lumii la ARN . [67] [68]
La începutul anilor 1970 , au fost descoperite retrovirusuri și transcriptază inversă , care au arătat pentru prima dată cum enzimele pot copia ARN în ADN (opusul căii obișnuite pentru transmiterea informațiilor genetice). Pentru această lucrare, David Baltimore , Renato Dulbecco și Howard Martin Temin au primit Premiul Nobel în 1975. În anul următor, Walter Fiers și echipa sa au determinat prima secvență de nucleotide a genomului complet al unui virus ARN, bacteriofagul MS2 . [69]
În 1977, intronii și îmbinarea ARN au fost descoperiți atât în virusurile mamiferelor, cât și în genele celulare, rezultând un premiu Nobel pentru Phillip Sharp și Richard Roberts în 1993. Primele molecule catalitice de ARN ( ribozime ) au fost identificate în primii ani 1980, aducând un Nobel Premiul acordat lui Thomas Cech și Sidney Altman în 1989. În 1990, genele care pot reduce la tăcere alte gene similare de plante au fost găsite în petunia , un proces cunoscut acum ca interferență ARN . [70] [71]
În același timp, s-au descoperit că ARN-urile lungi de 22 nt, numite acum microARN , au un rol în dezvoltarea Caenorhabditis elegans . [72] Studiile de interferență ARN au obținut un Premiu Nobel pentru Andrew Zachary Fire și Craig Cameron Mello în 2006 și un alt Premiu Nobel a fost acordat lui Roger Kornberg pentru studii de transcriere a ARN în același an. Descoperirea genelor ARN reglatoare a dus la încercări de a dezvolta medicamente produse cu ARN, cum ar fi siARN , pentru a reduce la tăcere genele. [73]
Notă
- ^ http://www.treccani.it/vocabolario/arn/
- ^ ARN: Molecula versatilă , pe learn.genetics.utah.edu , Universitatea din Utah , 2015.
- ^ Nucleotide și acizi nucleici ( PDF ). Chem.ucla.edu , Universitatea din California, Los Angeles . Adus la 13 octombrie 2015 (arhivat din original la 23 septembrie 2015) .
- ^ RN Shukla, Analiza cromozomilor , ISBN 978-93-84568-17-7 .
- ^ a b Berg JM, Tymoczko JL și Stryer L, Biochimie , 5th, WH Freeman and Company, 2002, pp. 118-19, 781-808, ISBN 0-7167-4684-0 ,OCLC 179705944 48055706 59502128 .
- ^ I. Tinoco și C. Bustamante, Cum se pliază ARN , în J. Mol. Biol. , vol. 293, nr. 2, 1999, pp. 271-281, DOI : 10.1006 / jmbi.1999.3001 , PMID 10550208 .
- ^ Higgs PG, structura secundară ARN: aspecte fizice și de calcul , în Revista trimestrială de biofizică , vol. 33, nr. 3, 2000, pp. 199-253, DOI : 10.1017 / S0033583500003620 , PMID 11191843 .
- ^ a b Nissen P, Hansen J, Ban N, Moore PB și Steitz TA, Baza structurală a activității ribozomilor în sinteza legăturilor peptidice , în Știința , vol. 289, nr. 5481, 2000, pp. 920-30, Bibcode : 2000Sci ... 289..920N , DOI : 10.1126 / science.289.5481.920 , PMID 10937990 .
- ^ a b Lee JC și Gutell RR, Diversitatea conformațiilor perechilor de baze și apariția lor în structura ARNr și motivele structurale ARN , în J. Mol. Biol. , vol. 344, n. 5, 2004, pp. 1225–49, DOI : 10.1016 / j.jmb . 2004.09.072 , PMID 15561141 .
- ^ Barciszewski J, Frederic B și Clark C, ARN biochimie și biotehnologie , Springer, 1999, pp. 73-87, ISBN 0-7923-5862-7 ,OCLC 52403776 .
- ^ Salazar M, Fedoroff OY, Miller JM, Ribeiro NS și Reid BR, Strand ADN în duplexurile hibride DNAoRNA nu este nici formă B, nici formă A în soluție , în Biochimie , vol. 32, nr. 16, 1992, pp. 4207–15, DOI : 10.1021 / bi00067a007 , PMID 7682844 .
- ^ Hermann T și Patel DJ, RNA bombează ca motive arhitecturale și de recunoaștere , în Structură , vol. 8, nr. 3, 2000, pp. R47 - R54, DOI : 10.1016 / S0969-2126 (00) 00110-6 , PMID 10745015 .
- ^ Mikkola S, Stenman E, Nurmi K, Yousefi-Salakdeh E, Strömberg R și Lönnberg H, Mecanismul scindării promovate de ionul metalic al legăturilor fosfodiesterului ARN implică o cataliză generală a acidului de către aquo ionul metalic la plecarea grupului părăsit , în tranzacțiile Perkin 2 , n. 8, 1999, pp. 1619-26, DOI : 10.1039 / a903691a .
- ^ Jankowski JAZ și Polak JM, Clinical gene analysis and manipulation: Tools, techniques and troubleshooting , Cambridge University Press, 1996, p. 14, ISBN 0-521-47896-0 ,OCLC 33838261 .
- ^ Yu Q și Morrow CD, Identificarea elementelor critice în tulpina acceptorului de ARNt și bucla TΨC necesare pentru infectivitatea virusului imunodeficienței umane de tip 1 , în J Virol , vol. 75, nr. 10, 2001, pp. 4902–6, DOI : 10.1128 / JVI.75.10.4902-4906.2001 , PMC 114245 , PMID 11312362 .
- ^ Elliott MS și Trewyn RW, biosinteza inozinei în ARN de transfer printr-o inserție enzimatică de hipoxantină , în J. Biol. Chem. , vol. 259, n. 4, 1983, pp. 2407-10, PMID 6365911 .
- ^ WA Cantara, Crain, PF, Rozenski, J, McCloskey, JA, Harris, KA, Zhang, X, Vendeix, FA, Fabris, D și Agris, PF,baza de date pentru modificarea ARN, actualizarea RNAMDB: 2011 , în Cercetarea Acizilor Nucleici , vol. 39, Ediția bazei de date, ianuarie 2011, pp. D195-201, DOI : 10.1093 / nar / gkq1028 , PMC 3013656 , PMID 21071406 .
- ^ Söll D și RajBhandary U, TRNA: Structură, biosinteză și funcție , ASM Press, 1995, p. 165, ISBN 1-55581-073-X ,OCLC 183036381 30663724 .
- ^ Kiss T, Modificare post-transcripțională a ARN-urilor celulare , ghidată de ARN nucleolar , în Jurnalul EMBO , vol. 20, nr. 14, 2001, pp. 3617–22, DOI : 10.1093 / emboj / 20.14.3617 , PMC 125535 , PMID 11447102 .
- ^ King TH, Liu B, McCully RR și Fournier MJ, structura și activitatea ribozomului sunt modificate în celulele lipsite de snoRNPs care formează pseudouridine în centrul peptidil transferazei , în Molecular Cell , vol. 11, n. 2, 2002, pp. 425–35, DOI : 10.1016 / S1097-2765 (03) 00040-6 , PMID 12620230 .
- ^ Mathews DH, Disney MD, Childs JL, Schroeder SJ, Zuker M și Turner DH, Incorporând constrângeri de modificare chimică într-un algoritm de programare dinamică pentru predicția structurii secundare ARN , în Proc. Natl. Acad. Sci. SUA , vol. 101, nr. 19, 2004, pp. 7287-92, bibcode : 2004PNAS..101.7287M , DOI : 10.1073 / pnas.0401799101 , PMC 409911 , PMID 15123812 .
- ^ Tan ZJ și Chen SJ,dependența de sare a stabilității acului nucleic , în Biophys. J. , voi. 95, nr. 2, 2008, pp. 738–52, Bibcode : 2008BpJ .... 95..738T , DOI : 10.1529 / biophysj.108.131524 , PMC 2440479 , PMID 18424500 .
- ^ Nudler E și Gottesman ME, Transcription termination and anti-termination in E. coli , în Genes to Cells , vol. 7, nr. 8, 2002, pp. 755–68, DOI : 10.1046 / j.1365-2443.2002.00563.x , PMID 12167155 .
- ^ Jeffrey L Hansen, Alexander M Long și Steve C Schultz, Structura ARN-polimerazei dependente de ARN a poliovirusului , în Structura , vol. 5, nr. 8, 1997, pp. 1109-22, DOI : 10.1016 / S0969-2126 (97) 00261-X , PMID 9309225 .
- ^ Ahlquist P, ARN polimeraze dependente de ARN, viruși și silențiere ARN , în Știință , vol. 296, nr. 5571, 2002, pp. 1270–73, Bibcode : 2002Sci ... 296.1270A , DOI : 10.1126 / science.1069132 , PMID 12016304 .
- ^ a b c Cooper GC și Hausman RE, The Cell: A Molecular Approach , 3rd, Sinauer, 2004, pp. 261-76, 297, 339-44, ISBN 0-87893-214-3 ,OCLC 174924833 52121379 52359301 56050609 .
- ^ Mattick JS și Gagen MJ, Evoluția rețelelor de gene multitask controlate: rolul intronilor și altor ARN-uri necodificatoare în dezvoltarea organismelor complexe , în Mol. Biol. Evol. , vol. 18, nr. 9, 1 septembrie 2001, pp. 1611–30, DOI : 10.1093 / oxfordjournals.molbev.a003951 , PMID 11504843 .
- ^ JS Mattick, ARN-uri necodificate: arhitecții complexității eucariote , în EMBO Reports , vol. 2, nr. 11, 2001, pp. 986–91, DOI : 10.1093 / embo-reports / kve230 , PMC 1084129 , PMID 11713189 .
- ^ Mattick JS, Provocarea dogmei: stratul ascuns de ARN-uri care nu codifică proteinele în organismele complexe ( PDF ), în BioEssays: News and Reviews in Molecular, Cellular and Developmental Biology , vol. 25, nr. 10, octombrie 2003, pp. 930–9, DOI : 10.1002 / bies.10332 , PMID 14505360 (arhivat din original la 6 martie 2009) .
- ^ Mattick JS, Programul genetic ascuns al organismelor complexe , în Scientific American , vol. 291, nr. 4, octombrie 2004, pp. 60–7, DOI : 10.1038 / scientificamerican1004-60 , PMID 15487671 (arhivat din original la 8 februarie 2015) .
- ^ a b c Wirta W, Mining the transcriptome – methods and applications , Stockholm, School of Biotechnology, Royal Institute of Technology, 2006, ISBN 91-7178-436-5 , OCLC 185406288 .
- ^ a b Rossi JJ, Ribozyme diagnostics comes of age , in Chemistry & Biology , vol. 11, n. 7, 2004, pp. 894–95, DOI : 10.1016/j.chembiol.2004.07.002 , PMID 15271347 .
- ^ Gueneau de Novoa P, Williams KP e Williams, The tmRNA website: reductive evolution of tmRNA in plastids and other endosymbionts , in Nucleic Acids Res , vol. 32, Database issue, 2004, pp. D104–8, DOI : 10.1093/nar/gkh102 , PMC 308836 , PMID 14681369 .
- ^ RW Carthew e Sontheimer, EJ., Origins and Mechanisms of miRNAs and siRNAs. , in Cell , DOI : 10.1016/j.cell.2009.01.035 , PMC 2675692 , PMID 19239886 . URL consultato il 20 febbraio 2009 .
- ^ Kung-Hao Liang e Yeh, Chau-Ting, A gene expression restriction network mediated by sense and antisense Alu sequences located on protein-coding messenger RNAs. , in BMC Genomics , DOI : 10.1186/1471-2164-14-325 , PMC 3655826 , PMID 23663499 . URL consultato l'11 maggio 2013 .
- ^ Wu L e Belasco JG, Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs , in Mol. Cell , vol. 29, n. 1, January 2008, pp. 1–7, DOI : 10.1016/j.molcel.2007.12.010 , PMID 18206964 .
- ^ Matzke MA e Matzke AJM, Planting the seeds of a new paradigm , in PLoS Biology , vol. 2, n. 5, 2004, pp. e133, DOI : 10.1371/journal.pbio.0020133 , PMC 406394 , PMID 15138502 .
- ^ Vazquez F, Vaucheret H, Rajagopalan R, Lepers C, Gasciolli V, Mallory AC, Hilbert J, Bartel DP e Crété P, Endogenous trans -acting siRNAs regulate the accumulation of Arabidopsis mRNAs , in Molecular Cell , vol. 16, n. 1, 2004, pp. 69–79, DOI : 10.1016/j.molcel.2004.09.028 , PMID 15469823 .
- ^ Watanabe T, Totoki Y, Toyoda A, Kaneda M, Satomi Kuramochi-Miyagawa, Yayoi Obata, Hatsune Chiba, Yuji Kohara, Tomohiro Kono, Toru Nakano, M. Azim Surani, Y Sakaki e H Sasaki, Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes , in Nature , vol. 453, n. 7194, May 2008, pp. 539–43, Bibcode : 2008Natur.453..539W , DOI : 10.1038/nature06908 , PMID 18404146 .
- ^ Sontheimer EJ e Carthew RW, Silence from within: endogenous siRNAs and miRNAs , in Cell , vol. 122, n. 1, July 2005, pp. 9–12, DOI : 10.1016/j.cell.2005.06.030 , PMID 16009127 .
- ^ Doran G, RNAi – Is one suffix sufficient? , in Journal of RNAi and Gene Silencing , vol. 3, n. 1, 2007, pp. 217–19 (archiviato dall' url originale il 16 luglio 2007) .
- ^ Pushparaj PN, Aarthi JJ, Kumar SD e Manikandan J,RNAi and RNAa — The Yin and Yang of RNAome , in Bioinformation , vol. 2, n. 6, 2008, pp. 235–7, DOI : 10.6026/97320630002235 , PMC 2258431 , PMID 18317570 .
- ^ Horwich MD, Li C, Matranga C, Vagin V, Farley G, Wang P e Zamore PD, The Drosophila RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC , in Current Biology , vol. 17, n. 14, 2007, pp. 1265–72, DOI : 10.1016/j.cub.2007.06.030 , PMID 17604629 .
- ^ Girard A, Sachidanandam R, Hannon GJ e Carmell MA, A germline-specific class of small RNAs binds mammalian Piwi proteins , in Nature , vol. 442, n. 7099, 2006, pp. 199–202, Bibcode : 2006Natur.442..199G , DOI : 10.1038/nature04917 , PMID 16751776 .
- ^ Horvath P e Barrangou R, CRISPR/Cas, the Immune System of Bacteria and Archaea , in Science , vol. 327, n. 5962, 2010, pp. 167–70, Bibcode : 2010Sci...327..167H , DOI : 10.1126/science.1179555 , PMID 20056882 .
- ^ Wagner EG, Altuvia S e Romby P, Antisense RNAs in bacteria and their genetic elements , in Adv Genet , Advances in Genetics, vol. 46, 2002, pp. 361–98, DOI : 10.1016/S0065-2660(02)46013-0 , ISBN 978-0-12-017646-5 , PMID 11931231 .
- ^ Gilbert SF, Developmental Biology , 7th, Sinauer, 2003, pp. 101 –3, ISBN 0-87893-258-5 , OCLC 154656422 154663147 174530692 177000492 177316159 51544170 54743254 59197768 61404850 66754122 .
- ^ Amaral PP e Mattick JS, Noncoding RNA in development , in Mammalian genome : official journal of the International Mammalian Genome Society , vol. 19, 7–8, October 2008, pp. 454–92, DOI : 10.1007/s00335-008-9136-7 , PMID 18839252 .
- ^ Heard E, Mongelard F, Arnaud D, Chureau C, Vourc'h C e Avner P, Human XIST yeast artificial chromosome transgenes show partial X inactivation center function in mouse embryonic stem cells , in Proc. Natl. Acad. Sci. USA , vol. 96, n. 12, 1999, pp. 6841–46, Bibcode : 1999PNAS...96.6841H , DOI : 10.1073/pnas.96.12.6841 , PMC 22003 , PMID 10359800 .
- ^ Steitz TA e Steitz JA, A general two-metal-ion mechanism for catalytic RNA , in Proc. Natl. Acad. Sci. USA , vol. 90, n. 14, 1993, pp. 6498–502, Bibcode : 1993PNAS...90.6498S , DOI : 10.1073/pnas.90.14.6498 , PMC 46959 , PMID 8341661 .
- ^ Xie J, Zhang M, Zhou T, Hua X, Tang L e Wu W,Sno/scaRNAbase: a curated database for small nucleolar RNAs and cajal body-specific RNAs , in Nucleic Acids Res , vol. 35, Database issue, 2007, pp. D183–7, DOI : 10.1093/nar/gkl873 , PMC 1669756 , PMID 17099227 .
- ^ Omer AD, Ziesche S, Decatur WA, Fournier MJ e Dennis PP, RNA-modifying machines in archaea , in Molecular Microbiology , vol. 48, n. 3, 2003, pp. 617–29, DOI : 10.1046/j.1365-2958.2003.03483.x , PMID 12694609 .
- ^ Cavaillé J, Nicoloso M e Bachellerie JP, Targeted ribose methylation of RNA in vivo directed by tailored antisense RNA guides , in Nature , vol. 383, n. 6602, 1996, pp. 732–5, Bibcode : 1996Natur.383..732C , DOI : 10.1038/383732a0 , PMID 8878486 .
- ^ Kiss-László Z, Henry Y, Bachellerie JP, Caizergues-Ferrer M e Kiss T, Site-specific ribose methylation of preribosomal RNA: a novel function for small nucleolar RNAs , in Cell , vol. 85, n. 7, 1996, pp. 1077–88, DOI : 10.1016/S0092-8674(00)81308-2 , PMID 8674114 .
- ^ Daròs JA, Elena SF e Flores R,Viroids: an Ariadne's thread into the RNA labyrinth , in EMBO Rep , vol. 7, n. 6, 2006, pp. 593–8, DOI : 10.1038/sj.embor.7400706 , PMC 1479586 , PMID 16741503 .
- ^ Kalendar R, Vicient CM, Peleg O, Anamthawat-Jonsson K, Bolshoy A e Schulman AH,Large retrotransposon derivatives: abundant, conserved but nonautonomous retroelements of barley and related genomes , in Genetics , vol. 166, n. 3, 2004, pp. 1437–50, DOI : 10.1534/genetics.166.3.1437 , PMC 1470764 , PMID 15082561 .
- ^ Podlevsky JD, Bley CJ, Omana RV, Qi X e Chen JJ,The telomerase database , in Nucleic Acids Res , vol. 36, Database issue, 2008, pp. D339–43, DOI : 10.1093/nar/gkm700 , PMC 2238860 , PMID 18073191 .
- ^ T. Blevins, R. Rajeswaran, PV. Shivaprasad, D. Beknazariants, A. Si-Ammour, HS. Park, F. Vazquez, D. Robertson e F. Meins,Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing , in Nucleic Acids Res , vol. 34, n. 21, 2006, pp. 6233–46, DOI : 10.1093/nar/gkl886 , PMC 1669714 , PMID 17090584 .
- ^ Jana S, Chakraborty C, Nandi S e Deb JK, RNA interference: potential therapeutic targets , in Appl. Microbiol. Biotechnol. , vol. 65, n. 6, 2004, pp. 649–57, DOI : 10.1007/s00253-004-1732-1 , PMID 15372214 .
- ^ Schultz U, Kaspers B e Staeheli P, The interferon system of non-mammalian vertebrates , in Dev. Comp. Immunol. , vol. 28, n. 5, 2004, pp. 499–508, DOI : 10.1016/j.dci.2003.09.009 , PMID 15062646 .
- ^ KA Whitehead, JE Dahlman, RS Langer e DG Anderson, Silencing or Stimulation? SiRNA Delivery and the Immune System , in Annual Review of Chemical and Biomolecular Engineering , vol. 2, 2011, pp. 77–96, DOI : 10.1146/annurev-chembioeng-061010-114133 , PMID 22432611 .
- ^ Dahm R, Friedrich Miescher and the discovery of DNA , in Developmental Biology , vol. 278, n. 2, 2005, pp. 274–88, DOI : 10.1016/j.ydbio.2004.11.028 , PMID 15680349 .
- ^ Caspersson T e Schultz J, Pentose nucleotides in the cytoplasm of growing tissues , in Nature , vol. 143, n. 3623, 1939, pp. 602–3, Bibcode : 1939Natur.143..602C , DOI : 10.1038/143602c0 .
- ^ Ochoa S, Enzymatic synthesis of ribonucleic acid ( PDF ), su Nobel Lecture , 1959.
- ^ Rich A e Davies, D, A New Two-Stranded Helical Structure: Polyadenylic Acid and Polyuridylic Acid , in Journal of the American Chemical Society , vol. 78, n. 14, 1956, pp. 3548–3549, DOI : 10.1021/ja01595a086 .
- ^ Holley RW, J. Apgar, GA Everett, JT Madison, M. Marquisee, SH Merrill, JR Penswick e A. Zamir, Structure of a ribonucleic acid , in Science , vol. 147, n. 3664, 1965, pp. 1462–65, Bibcode : 1965Sci...147.1462H , DOI : 10.1126/science.147.3664.1462 , PMID 14263761 .
- ^ Siebert S, Common sequence structure properties and stable regions in RNA secondary structures ( PDF ), su Dissertation, Albert-Ludwigs-Universität, Freiburg im Breisgau , 2006, p. 1 (archiviato dall' url originale il 9 marzo 2012) .
- ^ Szathmáry E, The origin of the genetic code: amino acids as cofactors in an RNA world , in Trends Genet , vol. 15, n. 6, 1999, pp. 223–9, DOI : 10.1016/S0168-9525(99)01730-8 , PMID 10354582 .
- ^ Fiers W, R. Ysebaert, F. Duerinck, G. Haegeman, D. Iserentant, J. Merregaert, W. Min Jou, F. Molemans, A. Raeymaekers, A. Van Den Berghe, G. Volckaert e M Ysebaert, Complete nucleotide-sequence of bacteriophage MS2-RNA: primary and secondary structure of replicase gene , in Nature , vol. 260, n. 5551, 1976, pp. 500–7, Bibcode : 1976Natur.260..500F , DOI : 10.1038/260500a0 , PMID 1264203 .
- ^ Napoli C, Lemieux C e Jorgensen R, Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans , in Plant Cell , vol. 2, n. 4, 1990, pp. 279–89, DOI : 10.1105/tpc.2.4.279 , PMC 159885 , PMID 12354959 .
- ^ Dafny-Yelin M, Chung SM, Frankman EL e Tzfira T,pSAT RNA interference vectors: a modular series for multiple gene down-regulation in plants , in Plant Physiol. , vol. 145, n. 4, December 2007, pp. 1272–81, DOI : 10.1104/pp.107.106062 , PMC 2151715 , PMID 17766396 .
- ^ Ruvkun G, Glimpses of a tiny RNA world , in Science , vol. 294, n. 5543, 2001, pp. 797–99, DOI : 10.1126/science.1066315 , PMID 11679654 .
- ^ Fichou Y e Férec C, The potential of oligonucleotides for therapeutic applications , in Trends in Biotechnology , vol. 24, n. 12, 2006, pp. 563–70, DOI : 10.1016/j.tibtech.2006.10.003 , PMID 17045686 .
Bibliografia
- David L. Nelson, Michael M. Cox, I Principi di Biochimica di Lehninger , 3ª ed., Bologna, Zanichelli , febbraio 2002, ISBN 88-08-09035-3 .
- ( EN ) RN Shukla, Analysis of Chromosomes , ISBN 978-93-84568-17-7 .
- ( EN ) Berg JM, Tymoczko JL e Stryer L, Biochemistry , 5th, WH Freeman and Company, 2002, ISBN 0-7167-4684-0 , OCLC 179705944 48055706 59502128 .
- ( EN ) Barciszewski J, Frederic B e Clark C, RNA biochemistry and biotechnology , Springer, 1999, ISBN 0-7923-5862-7 , OCLC 52403776 .
- ( EN ) Jankowski JAZ e Polak JM,Clinical gene analysis and manipulation: Tools, techniques and troubleshooting , Cambridge University Press, 1996, ISBN 0-521-47896-0 , OCLC 33838261 .
- ( EN ) Söll D e RajBhandary U,TRNA: Structure, biosynthesis, and function , ASM Press, 1995, ISBN 1-55581-073-X , OCLC 183036381 30663724 .
- ( EN ) Cooper GC e Hausman RE, The Cell: A Molecular Approach , 3rd, Sinauer, 2004, ISBN 0-87893-214-3 , OCLC 174924833 52121379 52359301 56050609 .
- ( EN ) Gilbert SF, Developmental Biology , 7th, Sinauer, 2003, ISBN 0-87893-258-5 , OCLC 154656422 154663147 174530692 177000492 177316159 51544170 54743254 59197768 61404850 66754122 .
- ( EN ) Wirta W, Mining the transcriptome – methods and applications , Stockholm, School of Biotechnology, Royal Institute of Technology, 2006, ISBN 91-7178-436-5 , OCLC 185406288 .
Voci correlate
Altri progetti
-
 Wikimedia Commons contiene immagini o altri file su RNA
Wikimedia Commons contiene immagini o altri file su RNA
Collegamenti esterni
- ( EN ) RNA , su Enciclopedia Britannica , Encyclopædia Britannica, Inc.
- RNA , in Dizionario di medicina , Istituto dell'Enciclopedia Italiana, 2010.
- ( EN ) IUPAC Gold Book, "ribonucleic acids (RNA)" , su goldbook.iupac.org .
- L'RNA ribosomico, sul portale RAI Scuola , su raiscuola.rai.it .
| Controllo di autorità | Thesaurus BNCF 25310 · LCCN ( EN ) sh85113850 · GND ( DE ) 4076759-0 · BNF ( FR ) cb12175223g (data) · BNE ( ES ) XX532187 (data) |
|---|







