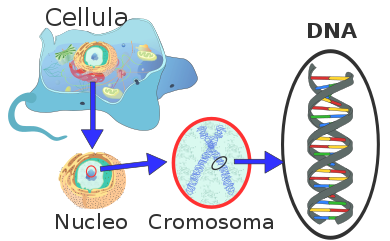
DNA
L „ de acid dezoxiribonucleic (ADN în abreviere, acidul dezoxiribonucleic engleza mai rar, în italiană, chiar și ADN [1] [2] [3] ) este un acid nucleic care conține informația genetică necesară pentru biosinteza de ARN și proteine , esențiale molecule pentru dezvoltarea și buna funcționare a majorității organismelor vii . [4]
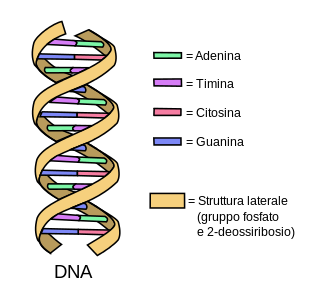
Din punct de vedere chimic, ADN - ul este un polimer cu lanț dublu organic a cărui monomeri sunt numite nucleotide (deoxiribonucleotide sau deoxiribonucleotide). Nucleotidele sunt formate din trei componente de bază: un grup fosfat , un zahar pentoză (dezoxiriboza, de asemenea , cunoscut sub numele de dezoxiriboză ) și o bază azotată care se leagă la dezoxiribozei cu o legătură N-glicozidică . Bazele azotate care intră în formarea de nucleotide sunt patru: adenina , timina , citozina și guanina (în ARN în loc de timină este " uracil ). ADN-ul poate fi definit global ca un lanț dublu polinucleotide (A, T, C, G), antiparalel, orientat, complementar, spiralized, informaționale.
Ordinea în dispunerea secvențială a nucleotidelor constituie informația genetică, care se traduce prin codul genetic în aminoacizii corespunzători. Secvența de aminoacizi a produs, a declarat polipeptida , formează proteina . Procesul de traducere genetică (denumit în mod obișnuit sinteza proteinelor ) este posibilă numai în prezența unei molecule intermediare de ARN , care este generat de complementaritate cu cele patru baze ale nucleotidelor ale ADN - ului într - un proces cunoscut sub numele de transcriere . Acest proces nu numai că generează ARN șuvițe destinate traducerii, dar , de asemenea , fragmente deja posibilitatea de a efectua mai multe funcții biologice ( de exemplu , în ribozomi , unde ARN - ul are o funcție structurală). Informația genetică este duplicat înainte de a diviziunii celulare prin replicarea ADN - ului , care este transmis în întregime în trecerea dintre generații diferite de celule.
In eukariote , ADN - ul este complexat în interiorul miezului în structuri numite cromozomi . In alte organisme, lipsit de un nucleu, poate fi organizat în cromozomi sau mai puțin (în bacterii este prezentă o singură moleculă de ADN circular dublu catenar, în timp ce virusurile pot avea genomuri la ADN sau ARN ). In cadrul cromozomi, proteinele din cromatinei ca histonelor , coesine și condensine, organizează DNA și înveliți în structuri ordonate. Aceste structuri conduc interacțiunea dintre codul genetic și proteinele responsabile de transcriere, care contribuie la controlul transcripției genei.
fundal
ADN - ul a fost izolat inițial dintr - un biochimic elvețian Friedrich Miescher , care, în 1869 , a identificat o substanță microscopice în puroi de bandaje chirurgicale folosite. Deoarece această moleculă a avut poziția sa în miez , el a numit nuclein. [5] În 1919 Phoebus Levene a identificat structura nucleotida, compusă din baze azotate, zahăr și fosfat. [6] Levene a sugerat că ADN - ul a constat dintr - un fir de nucleotide legate între ele prin intermediul fosfatului. Cu toate acestea, el a fost convins că acest filament era scurt și că bazele au fost aranjate într-o ordine precisă repetată. In 1937 William Astbury a prezentat primele rezultate ale unor studii de difracție de raze X , care a arătat că ADN - ul are o structură foarte regulată [7] . În 1944 Erwin Schrödinger a afirmat că, din moment ce în conformitate cu sistemele fizicii cuantice de cativa atomi au un comportament dezordonat, materialul genetic trebuie să constea dintr - o molecula mare non-repetitive, suficient de stabil pentru a menține informația genetică, numită „cristal aperiodic“. [8]
In 1928 Frederick Griffith a descoperit că personajele de forma buna ( „netede“) de pneumococ ar putea fi transferate la forma brute ( „șifonate“), prin amestecarea resturile de bacterii netede dur cu bacterii moarte în viață. [9] Acest sistem, oferind în același timp nici o dovada a ceea ce a fost substanța care a determinat modificarea, a arătat că ceva ar putea transporta informații genetice din resturile de bacterii moarte la cele vii. Apoi , el a vorbit despre un principiu de transformare capabile să modifice bacteriile vii. În 1943 Oswald Avery a arătat într - un experiment celebru cu Colin MacLeod si Maclyn McCarty ca ADN - ul este principiul care stă la baza acestui fenomen de transformare. [10] Rolul ADN - ului în " moștenire a fost încercat, în cele din urmă, în 1953 de către Alfred Hershey și Martha Chase printr - un alt experiment clasic , care a arătat că materialul genetic al T2 fagului este de fapt ADN - ul. [11]
1953 a fost și anul în care, prin imagini de difracție cu raze X suplimentare [12] luate de Rosalind Franklin , chimie fizică engleză, James Watson și Francis Crick au prezentat [12] , în revista Nature , cea care a fost acum stabilit ca primul model precis al structurii ADN - ului, [13] sau modelul dublu helix. În desen comp a fost Odile Crick , soția pictorului și Crick. Dovezile experimentale de susținere a modelului Watson și Crick a fost raportată într-o serie de cinci articole publicate in aceeasi problema a Naturii. [14] Acestea au inclus articolul lui Franklin și Raymond Gosling , care conținea datele de difracție cu raze X, critice pentru a sprijini modelul. [15] [16] Acest număr conținea , de asemenea , un articol cu privire la structura ADN - ului scris de Maurice Wilkins . [17] În 1962 , după moartea lui Rosalind Franklin ( din cauza unui cancer cauzat, probabil, de la doze mari de raze X la care a fost expus în cursul experimentelor sale), Watson, Crick și Wilkins au primit în comun Premiul Nobel pentru medicină . [18] De la descoperirea modelului sa bazat în principal pe datele de Rosalind Franklin, chiar și astăzi există opinii foarte diferite în cadrul comunității științifice peste care ar trebui să primească premiul.
Într - o prezentare importantă în 1957 , Crick a propus dogma centrală a biologiei moleculare , stabilirea relației dintre ADN, ARN și proteine. [19] Confirmarea finală a mecanismului de replicare bazat pe structura de dublu helix a fost furnizată în 1958 de către " Meselson-Stahl . [20] O lucrare mai târziu de Crick a demonstrat cum codul genetic a fost bazat pe tripleți nu se suprapun de baze, care sa permita Har Gobind Khorana , Robert Holley și Marshall Warren Nirenberg să descifreze. [21] Aceste descoperiri sunt baza moderne biologiei moleculare .
In 1961 Marshall Nirenberg și Severo Ochoa a descoperit că fiecare triplet de nucleotide care codifică pentru un anumit aminoacid.
Structura
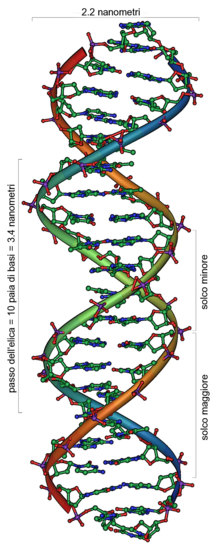
ADN - ul este un lung polimer format din unități repetitive de nucleotide . [22] [23] Lanțul ADN este mare între 22 și 26 Ångström ( de la 2,2 la 2,6 nanometri ) și fiecare unitate nucleotidică este lung 3.3 Angstrom (0,33 nm). [24] Deși fiecare unitate ocupă un spațiu foarte mic, lungimea polimerilor ADN pot fi surprinzător de mare, deoarece fiecare filament poate conține mai multe milioane de nucleotide. De exemplu, cel mai mare cromozom uman (pe cromozomul 1 ) conține aproape 250 de milioane de perechi de baze . [25]
In organismele vii, ADN-ul este aproape niciodată prezentă sub forma unui singur fir, ci ca o pereche de fire ferm asociate una cu cealaltă. [13] [26] Acestea sunt interconectate pentru a forma o structură helix dublu definită. Fiecare nucleotidă constă dintr - un schelet lateral, care permite să se lege covalent cu nucleotide adiacente, și o bază azotată, care stabilește legături de hidrogen cu corespunzătoare azotoase de bază prezentă pe filament opus. Compusul format dintr - o bază azotată legată de zahăr este definit nucleozida ; o nucleotidă este un nucleozid în schimb la care sunt legați unul sau mai multe grupări fosfat. [27]
Structura laterală a ADN - ului este compusă din unități repetate și alternante de grupări fosfat și 2-dezoxiriboză, [28] o pentoză zahăr (cu cinci atomi de carbon atomi ) care se leagă la fosfați adiacente prin legături fosfodiesterice la al treilea și al cincilea carbon; în practică, fiecare moleculă fosfat formează o punte de legătură moleculară, prin legături phosphodiesteric, carbonul din "poziția unei molecule dezoxiriboză cu care în 5 '3 poziția următoare zahăr. Consecința acestor legături asimetrice este aceea că fiecare fir de ADN are un sens, determinat de direcția legăturilor fosfodiesterice. Bazele azotate, pe de altă parte, se alăture în poziția 1“a zahărului deoxiriboză cu legături N-glicozidice. Într-un dublu helix, direcția de o catenă este opusă celei a catenei complementare. Din acest motiv, cele două filamente care alcătuiesc un dublu helix sunt numite antiparalel. Capetele asimetrice ale catenei de ADN sunt definite capetele 5 „ (cinci prime) și capetele 3“ ( primii trei). Diferența principală dintre ADN și ARN este pentose zahărul utilizat: ARN, de fapt, foloseste riboză . [26]
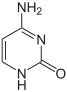
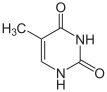
Dublu helix a ADN-ului este stabilizată prin legăturile de hidrogen care se stabilesc între bazele azotate prezente pe cele două componente. [29] Cele patru baze , care sunt prezente în ADN sunt " adenina (prescurtat cu litera A), citozină (C), guanina (G) și timina (T). Toate cele patru baze au o structură heterociclică , dar adenină și guanină sunt, din punct de vedere structural, derivați ai purinei și , prin urmare , a spus baze purinice, în timp ce citozină și timină sunt legate de pirimidinic și a spus bazele pirimidinice. [26] Există o bază cincea, tip pirimidinic, numit uracil (U), dar nu este de obicei prezent în lanțurile ADN. Uracil este de asemenea prezentă în catenele de ARN în loc de timină, de care diferă în lipsa unui grup de metil . Uracil este prezentă în ADN-ul numai ca un produs de degradare a citozinei. Numai în bacteriofag PBS1 această bază poate fi utilizată în cadrul DNA. [30] Dimpotrivă, este mult mai frecventă a localiza în interiorul timina de molecule de ARN, din cauza metilare enzimei de diferite uracili. Acest eveniment are loc , de obicei , în detrimentul ARN cu funcție structurală sau enzimatică ( ARNr și ARNt ). [31]
Helix dublu este o spirală dreptaci. Deoarece cele două filamente înșurubați ele însele, caneluri între diferitele grupuri de fosfat rămân expuse. Șanțul major este de 22 Â larg, în timp ce minor canelură 12 Â larg. [32] Diferitele amplitudinile celor două caneluri se traduce concret într - o altă accesibilitate a bazelor, în funcție de faptul dacă acestea sunt în canelura majoră sau minoră. Proteinele de legare de ADN, cum ar fi factorii de transcripție, prin urmare, de obicei, face contact cu bazele prezente în majore santul. [33] [34]
Asociere de bază
Fiecare tip de bază prezente pe un singur fir formează o legătură cu baza plasată pe catena opusă. Acest eveniment este cunoscut sub numele de împerechere complementare . Bazele purinice formează legături de hidrogen cu baze pirimidinice: A poate doar lega T și G poate numai bind C. Asocierea celor două baze este denumită în mod obișnuit de bază pereche și este cel mai frecvent utilizate unitatea de măsură pentru a defini lungimea unei molecule de ADN. Deoarece legăturile de hidrogen nu sunt covalente , ele pot fi rupte și asamblate într - un mod relativ simplu, deoarece acestea sunt legături de energie înaltă. Cele două filamente pot fi deplasate departe unul de altul, așa cum se întâmplă pentru o balama , atât de ridicate temperaturi care prin acțiunea mecanică (cum se produce în cursul replicarea ADN - ului ). [35] Consecința acestei complementarități este că toate informațiile conținute în dublu helix pot fi duplicate de pe ambele catene, un eveniment fundamental pentru replicarea ADN - ului corect. [22]
Cele două tipuri de perechi de baze formează un număr diferit de legături de hidrogen: A și T formă de două, G și C trei. Din acest motiv, stabilitatea legăturii GC este mult mai mare decât cel de la. În consecință, stabilitatea generală a unei molecule de ADN este direct legată de frecvența GC prezentă în molecula în sine, precum și lungimea helixului: o moleculă de ADN, prin urmare, este cu atât mai stabilă cu cât conține GC și este lung . [36] O altă consecință a acestui eveniment este faptul că regiunile de ADN care trebuie să fie ușor separate conțin o concentrație ridicată de A și T, așa cum se întâmplă , de exemplu , pentru cutia Pribnow de bacterii promotori , a căror secvență este de fapt TATAAT. [37]
În laborator, stabilitatea interacțiunii dintre filamentele este măsurată prin temperatura necesară pentru a rupe toate legăturile de hidrogen, denumit temperatura de topire (sau T m). Când toate legăturile de hidrogen sunt rupte, toroanele individuale diferite și pot lua pe structuri foarte variat. [38]
Stabilizarea dublu helix, în orice caz, nu se datorează numai legături de hidrogen, dar , de asemenea , la interacțiuni hidrofobe și stivuire pi . [39]
Sens și antisens

O secvență ADN este definită mod dacă secvența sa este aceeași ca și corespunzătoare mARN . Secvența de pe catena opusă este în locul numit antisens. Deoarece ARN polimeraza lucrare care produce o copie complementară, filamentul necesară pentru transcriere este antisens. Atât în procariote și eucariote sunt produse numeroase molecule antisens de ARN pornind de la secvențele senzoriale. Funcția acestor non-codare RNAs nu a fost încă pe deplin elucidat. [40] Se crede că ARN - ul antisens poate juca un rol în a „reglarea expresiei genelor . [41]
Există anumite secvențe de ADN, atât în procariote și eucariotelor (dar mai ales în plasmide și virusuri ) , în care diferența dintre secvențele de sens și antisens este mai puțin clară, deoarece secvențele de anumite gene se suprapun reciproc. [42] În aceste cazuri, prin urmare, unele secvențe joacă o sarcină dublă: pentru a codifica o proteină atunci când sunt citite în direcția 5“ → 3 „pe un singur fir; codifica o alta, dacă citiți pe de altă parte (întotdeauna în direcția 5“ → 3 „). In bacterii, această suprapunere gena este adesea implicată în reglarea transcrierii, [43] în timp ce în virus fenomenul se datorează necesității de a conține într - un mic genom o cantitate mare de informații. [44] Un alt mod de a reduce dimensiunea genomului este identificat de alte virusuri, care conțin molecule liniare sau circulare ADN mono-catenar. [45] [46]
supercoiling
ADN - ul poate fi distorsionat așa cum este cazul pentru o frânghie printr - un proces numit supercoiling . Atunci când ADN - ul este într - o stare relaxată, un filament de-a lungul unui viraj complet în jurul axei fiecare 10.4 perechi de baze. Pe de altă parte, în cazul în care ADN-ul este distorsionat, numărul de baze poate crește sau descrește. [47] Starea de supercoiling este situat într - o moleculă de ADN este definită topologie . Dacă ADN - ul este învelit în direcția helix, aceasta se numește supercoiling pozitiv, cu bazele strânse împreună într - un mod mai marcat. În caz contrar, este numit supercoiling negativ. In natura, cele mai multe dintre moleculele de ADN au o usoara supercoiling negativa, introdus de enzime definite topoizomeraza . [48] Aceste enzime sunt necesare și în procese , cum ar fi transcrierea și replicarea ADN - ului, deoarece acestea sunt în măsură să rezolve stresul topologic indus prin aceleași procese. [49]
Structuri dublu helix alternative
ADN - ul există în diferite conformații . Ele sunt numite A-ADN, B-ADN, C-ADN, D-ADN, [50] E-ADN, [51] H-ADN, [52] L-ADN, [50] P-ADN [53] și Z-ADN. [28] [54] În orice caz, conformations A-ADN, B-ADN și Z-ADN au fost observate numai în sistemele biologice naturale. Conformația ADN - ului poate depinde de secvența, de supraspiralare, prin prezența modificărilor chimice ale bazelor solventului sau prin condiții , cum ar fi concentrația ionilor de metale . [55] Dintre aceste conformații, forma B este cea mai frecventă în condițiile standard ale celulelor. [56] Cele două conformații alternative sunt diferite din punct de vedere al geometriei și dimensiunilor.
Forma A este lată în sens orar spirala (minor canelura este largă , dar superficial, cu atât mai mare este mai îngust și adânc), cu un pas de 2,9 nm (aproximativ 11BP), și un diametru de 2,5 nm. Această conformație este prezentă în condiții non-fiziologice, când ADN-ul este deshidratat. În condiții fiziologice, această caracterizează conformația ADN - ului și ARN - ului heteroduplex și complexul format de asociații ADN-proteină. [57] [58]
Z conformație este tipic în locul secvențelor care prezintă modificări chimice , cum ar fi de metilare și a întinderile de ADN bogate în baze C și G. Aceasta presupune o tendință stângaci, vizavi de conformația B. [59] Are pas de 4,6 nm și un diametru de 1,8 nm, majore Fisura mai superficial și minor una mai îngustă; își datorează numele de model în zig-zag pe care-l caracterizează. Aceste structuri neobișnuite pot fi recunoscute de specific Z- proteine de legare de ADN , cu consecințe semnificative în reglarea transcripției, deși nu este în natura exemplelor au fost găsite. [60]
Structuri alternative la dublu helix
Terminalul regiuni ale cromozomilor secvențe liniare sunt repetate a spus telomere . Funcția principală a acestor regiuni este de a permite celulei de a reproduce capetele cromozomilor fără nici o pierdere de informație genetică, deoarece polimerazele ADN implicate în replicarea ADN - ului sunt în imposibilitatea de a reproduce capetele 3“ale cromozomilor.. [62] În cazul în care un telomerii de cromozomi care nu a avut, de fapt, ea devine un pic mai scurt , cu fiecare replicare, cu riscul de a pierde secvențe de codificare. Printr - un anumit tip de ADN polimerază (numite telomerazei ), cu toate acestea, telomerii menține constant lungimea lor, protejând astfel partea interioară a cromozomului. In celulele umane, telomerii sunt compuse din mai multe mii de repetări ale unei secvențe simplu formată din TTAGGG. [63]
Această secvență guanină bogat poate stabiliza capetele de cromozomi formând structuri neobișnuite, compuse din unități de patru baze azotate în locul celor canonice două. Acest lucru se datorează interacțiunii între patru guanina, care formează o structură planară care stive deasupra altor structuri de același tip, pentru a obține un filament stabil definit G-cvadruplex structura. [64] Aceste structuri sunt stabilizate prin formarea de legături de hidrogen , care se stabilesc între vârfurile și bazele de chelatizare cu un ion metalic, amplasat în centrul fiecăreia dintre cele patru unități de baze. [65]
În plus față de acestea, telomerii generează , de asemenea , structuri circulare, numite telomerilor-bucle sau T-bucle. În acest caz, firul unic de ADN se pliază pentru a forma cercuri mari, stabilizate de proteine specifice care leagă telomerii. [66] La sfârșitul buclei T, ADN - ul monocatenar face contact cu un dublu catenară, care se deschide și formează o structură în triplu helix . Această structură este numită buclă de deplasare sau D-bucla. [64]
modificări chimice
modificări de bază
L ' expresia genelor unui anumit locus este influențat de structura pe care cromatinei presupune în același locus. Regiunile eterocromatiniche (caracterizate prin puțin sau deloc expresie) sunt pe larg metilat pe citozina . Metilarea citozină, de exemplu, este crucial pentru " inactivarea cromozomului X . [67] Nivelul mediu de metilare este foarte variabilă între diferitele organisme: Caenorhabditis elegans nu prezintă metilare citozina, in timp ce vertebrate prezintă niveluri mai ridicate, cu aproximativ 1% din genomul care conține 5-metilcitozină. [68] 5-metilcitozină, fiind susceptibile la dezaminare spontană, are o bază la care incidența mutațiilor este foarte mare. [69]
Modificarile ulterioare ale bazelor sunt de metilare adenină (prezente în bacterii) și glicozilarea uracil baze care produce așa-numitul J din kinetoplastids . [70] [71]
Deteriorarea ADN-ului

ADN - ul poate fi modificat prin acțiunea numeroși agenți, definite generic mutageni ; este esențial să se nota, totuși, că o mutație - care este o schimbare rara, aleatoare, care modifică secvența de baze azotate - nu este neapărat un eveniment pernicioasă, ci mai degrabă este la baza evoluției: mutația menționată mai sus trebuie totuși să drumul său în rețeaua densă cibernetica celulară , cât și în mediul în care organismul viu în viața de întrebare și funcționează; în cazul în care sunt depășite aceste puncte de restricție (foarte selectiv având în vedere complexitatea lor intrinsecă, marea majoritate a mutațiilor, de fapt, se dovedesc a nu fi avantajos sau chiar neutru), un organism îmbogățit de mutație va fi obținută. Agenții Alterand includ, de exemplu, oxidanți , agenți de alchilare și , de asemenea , de înaltă energie de radiație , cum ar fi razele X și UV raze .
Tipul de daune cauzate ADN - ului depinde de tipul de agent: UV, de exemplu, care afectează ADN-generând formarea dimerilor timină , constând din poduri aberante ce se stabilesc între bazele pirimidinice adiacente. [73] agenți de oxidare cum ar fi radicalii liberi sau peroxid de hidrogen , în schimb, ele produc mai multe daune tip eterogen, cum ar fi modificări ale bazelor (în particular guanină) sau ADN dublu-strand pauze. [74] Conform mai multor studii, în fiecare celulă umană cel puțin 500 de baze pe zi sunt supuse degradării oxidative. [75] [76] Dintre aceste leziuni, cele mai periculoase sunt dublu catenare pauze, deoarece aceste daune sunt cel mai greu de reparație și constituie sursa primară a mutațiilor punctiforme și decalare care se acumulează pe secvențe genomice, precum translocare cromozomul . [77]
Mulți agenți au capacitatea lor mutagen de intercalarea între două nucleobaze consecutive. Moleculele de intercalare sunt în mod tipic plane și aromatice , cum ar fi etidium , daunomicina , doxorubicina sau talidomida . Pentru un element de intercalare pentru a găsi un loc între cele două baze, dublu helix trebuie să se deschidă și să piardă conformația standard. Aceste modificări structurale inhibă atât transcripție și replicarea ADN și crește posibilitatea de mutații. Din acest motiv, moleculele de intercalare sunt considerate cancerigene , așa cum sa demonstrat prin numeroase studii pe molecule , cum ar fi Benzopirina „l acridina , l“ aflatoxinei și bromură de etidiu . [78] [79] [80] În orice caz, datorită capacității lor de a inhiba transcriere și replicare, aceste molecule sunt de asemenea folosite in chimioterapie pentru a inhiba creșterea rapidă a celulelor neoplazice. [81]
Dispunerea ADN
In eucariotelor , ADN - ul este de obicei prezent în termen de cromozomi liniari (circular în procariote ). Suma tuturor cromozomii unei celule este sa genom ; Genomului uman are aproximativ 3 miliarde de perechi de baze continute in 46 de cromozomi. [82]
Dispoziția finală în cromozomi urmează reguli precise ierarhice de ambalaje. In celulele, de fapt, ADN - ul dublu-catenar nu pot fi aranjate la întâmplare, dar trebuie să respecte reguli precise de sortare. Aceste măsuri sunt necesare, deoarece lungimea componentelor ADN-ului este de obicei foarte mare și ar crea probleme serioase pentru celula gazdă. De exemplu, cromozomul Escherichia coli , cel mai studiat procariot din istoria biomedicinei, măsurând aproximativ 1 mm. Într-o celulă numai 2 um lung, cum ar fi cea de E. coli, aranjamentul aleatoriu a unui astfel de cromozomi o poate genera probleme. În cazul în care o moleculă de această lungime au fost aranjate aleatoriu, de fapt, o celula ar fi nevoie de cel puțin 1000 de ori mai mare. Metodele de ambalare sunt diferite între organisme procariote și eucariote .
Procariote
In cele mai multe celule bacteriene ADN - ul este dispus pe un singur cromozom circular (și are, ca și multe alte bacterii, o singură origine de replicare ), așa cum a prezis de numeroase experimente de legătură și în final , materializate în celulele cultivate cu tritium- etichetate timina. .
Mecanismele implementate de celula procariotă pentru a reduce spațiul necesar constă mai întâi în mascarea sarcinile negative prezente pe ADN - ul prin asocierea sa cu poliamine, încărcate pozitiv, cum ar fi spermină și spermidină . În plus față de acestea, ADN-ul procariote, de asemenea, face contact cu numeroase proteine mici, care compact structura generală a ADN-ului. Printre ei, figura H-NS , un dimer cu funcții foarte similare cu histonelor eucariote. În fiecare celulă de E. coli sunt în medie 20.000 de molecule de H-NS, care sunt aranjate de-a lungul ADN-ului, la o distanță de aproximativ 400 pb.
Il DNA di E.coli è inoltre molto superavvolto . Tale fenomeno contribuisce ulteriormente al compattamento del DNA, permettendo ad esso di disporsi comodamente all'interno della cellula.
Eucarioti
Negli eucarioti l'impacchettamento è ottenuto attraverso diversi accorgimenti. Il DNA è associato ad un gran numero di proteine: l'associazione complessiva DNA-proteine è definita cromatina , la cui struttura è ampiamente conservata tra tutti gli organismi eucarioti.
Le proteine cromatiniche più abbondanti sono gli istoni , una famiglia di polipeptidi basici presenti nel nucleo. Le principali proteine istoniche sono H1 , H2A , H2B , H3 e H4. La basicità degli istoni è dovuta alla grande quantità di amminoacidi carichi positivamente ( lisina e arginina ), in grado di instaurare interazioni elettrostatiche con i gruppi fosfato del DNA. Le proteine istoniche sono anche pesantemente modificate, proprio sui residui carichi, da modificazioni post-traduzionali , tra cui l'aggiunta di acetili , di fosfati e di metili , che neutralizzano la carica positiva o la rendono negativa.
Le sequenze amminoacidiche di quattro dei cinque istoni (H2A, H2B, H3 e H4) sono altamente conservate, anche tra specie molto diverse. La sequenza di H1 presenta invece maggiori variazioni lungo l'evoluzione: in alcuni organismi, H1 non è nemmeno presente in tutti i tessuti (ad esempio negli eritrociti degli uccelli H1 è sostituita da un sesto istone, chiamato H5). La presenza di differenti H1, in ogni caso, non modifica sostanzialmente la struttura complessiva dell'apparato istonico (definito nucleosoma ), che resta ampiamente conservato nell'architettura nella quasi totalità degli eucarioti.
Funzioni biologiche
Nel genoma, l'informazione è conservata in sequenze di DNA chiamate geni . La trasmissione dell'informazione contenuta nei geni è garantita dalla presenza di sequenze di basi azotate complementari. Infatti, durante la trascrizione , l'informazione può essere facilmente copiata in un filamento complementare di RNA. Solitamente, tale copia di RNA è utilizzata per sintetizzare una proteina, attraverso un processo definito traduzione (o sintesi proteica). In alternativa, una cellula può semplicemente duplicare l'informazione genetica attraverso un processo definito replicazione del DNA .
Struttura del genoma
Negli organismi eucarioti, il DNA genomico è localizzato all'interno del nucleo cellulare, nonché in piccole quantità all'interno di mitocondri e cloroplasti . Nei procarioti, il DNA è invece racchiuso in un organello irregolare, privo di membrana, contenuto nel citoplasma, chiamato nucleoide . [83] L'informazione è contenuta all'interno dei geni, unità ereditarie in grado di influire sul fenotipo dell'organismo. Ogni gene contiene un open reading frame (regione in grado di essere trascritta a RNA) e una regione regolatoria, costituita sia da un promotore che da enhancers .
In molte specie , solo una piccola frazione della sequenza totale di un genoma può essere trascritta e tradotta. Ad esempio, solo l'1,5% del genoma umano è costituito da esoni codificanti una proteina, mentre più del 50% consiste di sequenze ripetute di DNA non codificante . [84] La ragione per cui ci sia una tale quantità di DNA non codificante non è tuttora completamente chiara ed è stata definita come enigma del C-value . [85] In ogni caso, le sequenze di DNA che non codificano una proteina possono essere trascritte in RNA non codificante , coinvolto nella regolazione dell' espressione genica . [86]

Alcune sequenze non codificanti ricoprono un ruolo strutturale per i cromosomi. Le regioni telomeriche e centromeriche contengono solitamente pochissimi geni, ma sono necessarie per la funzione e la stabilità dei cromosomi. [88] Nell'uomo, grandi quantità di DNA non codificante si ritrovano negli pseudogeni , copie di geni rese inattive dalla presenza di una mutazione. [89] Queste sequenze sono considerate come fossili molecolari, anche se esistono evidenze secondo le quali si può ipotizzare che siano una sorta di materiale grezzo necessario per la creazione di nuovi geni attraverso i processi di duplicazione genica e di evoluzione divergente . [90]
Trascrizione e traduzione
Un gene è una sequenza di DNA che contiene le informazioni in grado di influire sulle caratteristiche del fenotipo dell'organismo. All'interno di un gene, la sequenza di basi di DNA è utilizzata come stampo per la sintesi di una molecola di RNA che, nella maggior parte dei casi, è tradotta in una molecola peptidica.
Il meccanismo attraverso il quale la sequenza nucleotidica di un gene è copiata in un filamento di RNA è detto trascrizione ed avviene per mezzo dell'enzima RNA polimerasi . Il filamento di RNA può andare incontro a destini differenti: alcune molecole di RNA hanno funzioni di tipo strutturale (come quelle che si trovano all'interno del ribosoma ) o catalitica (molecole note come ribozimi ); la funzione più nota è tuttavia quella della traduzione in proteine tramite la produzione mRNA . Il processo di traduzione avviene nel citoplasma, dove gli mRNA si associano ai ribosomi, ed è mediato dal codice genetico . Il ribosoma permette la lettura sequenziale dei codoni del mRNA, favorendone il riconoscimento e l'interazione con specifici tRNA , molecole che trasportano gli amminoacidi corrispondenti ad ogni singolo codone.
Il codice genetico
Il codice genetico consiste di parole di tre lettere chiamate codoni , costituite dalla sequenza di tre nucleotidi (ad esempio ACU, CAG, UUU), presenti sull' mRNA , ognuna delle quali è associata ad un particolare amminoacido. Ad esempio la timina ripetuta in una serie di tre (UUU) codifica la fenilalanina . Utilizzando gruppi di tre lettere si possono avere fino a 64 combinazioni diverse ( ), in grado di codificare i venti diversi amminoacidi esistenti. Poiché esistono 64 triplette possibili e solo 20 amminoacidi, il codice genetico è detto ridondante (o degenere ): alcuni amminoacidi possono infatti essere codificati da più triplette diverse. Non è invece vero il contrario: ad ogni tripletta corrisponderà un solo amminoacido (senza possibilità di ambiguità). Esistono infine tre triplette che non codificano alcun amminoacido, ma rappresentano codoni di stop (o nonsense ), ovvero indicano il punto in cui, all'interno del gene, termina la sequenza che codifica la proteina corrispondente: si tratta dei codoni UAA, UGA e UAG.
Replicazione

La divisione cellulare, necessaria ad un organismo per crescere, richiede una duplicazione del DNA cellulare, in modo che le cellule figlie possano avere la stessa informazione genetica della cellula madre. La struttura a doppia elica del DNA permette un meccanismo estremamente semplice per la replicazione del DNA. I due filamenti, infatti, sono separati e da ognuno viene creato un filamento complementare, ad opera di un enzima chiamato DNA polimerasi . Con questo meccanismo, le basi presenti sul filamento figlio sono determinate da quelle presenti sul filamento parentale: è proprio attraverso questo meccanismo che le cellule figlie presentano genoma identico alla cellula madre (salvo errori avvenuti durante il processo, che portano alla comparsa di mutazioni). Tale tipo di replicazione, che porta a doppie eliche costituite da un filamento preesistente e uno neoformato è detta semiconservativa .
Per iniziare la replicazione, occorre anzitutto l'apertura della forca replicativa , attraverso la parziale denaturazione del DNA a doppia elica, portata a termine dalle elicasi e dalle single-strand-binding proteins (SSBPs): le elicasi sono enzimi che separano attivamente i due filamenti usando l'energia dell' ATP ; le SSBPs sono in grado di mantenere la denaturazione del DNA legandosi esclusivamente alle porzioni a singolo filamento e stabilizzandole. Nelle molecole di DNA circolari dei procarioti si ha una sola regione di origine della replicazione dalla quale partono due forche replicative (la struttura prende il nome di bolla di replicazione ). Quando le due forche si incontrano dal lato opposto la replicazione è completata. Negli eucarioti la replicazione di ogni cromosoma inizia invece in più punti.
Le DNA polimerasi , enzimi capaci di costruire una nuova catena solo in direzione 5'-3', sono stati individuati per la prima volta da Arthur Kornberg , il quale, grazie ad un suo famoso esperimento, [91] identificò la DNA polimerasi I in Escherichia coli . La reazione della DNA polimerasi è diretta dallo stampo , perché produce un nuovo filamento di DNA esattamente complementare ad uno preesistente che funge, appunto, da stampo. La DNA polimerasi non è in grado di iniziare la sintesi di un filamento ex novo , mentre può allungare un filamento polinucleotidico preesistente. In una cellula in replicazione, dunque, è indispensabile la presenza di un filamento preesistente (detto primer ), che consiste solitamente in un breve segmento di RNA complementare allo stampo, sintetizzato da enzimi specifici detti primasi .
Dal momento che le DNA polimerasi sono in grado di svolgere la loro attività solo in direzione 5'-3', esse hanno messo a punto diversi meccanismi per copiare i due filamenti della doppia elica. [92] Un filamento (chiamato filamento guida ) può essere replicato in modo quasi continuo, man mano che viene esposto, l'altro ( filamento lento ) risulta invece disseminato da brevi filamenti di DNA di nuova sintesi (i frammenti di Okazaki ), ognuno dei quali presenta un innesco iniziale di RNA. I nuovi filamenti devono essere quindi completati mediante la rimozione degli inneschi da parte di endonucleasi e il riempimento degli spazi rimasti ad opera di polimerasi di riparazione . Successivamente tutti questi frammenti di DNA di nuova sintesi del filamento lento vengono legati dalle DNA ligasi .
Interazioni con proteine
Tutte le funzioni del DNA dipendono dalle sue interazioni con specifiche proteine. Tali interazioni possono sia essere aspecifiche, sia prevedere un legame estremamente specifico della proteina a una singola sequenza di DNA. Sono numerosi anche gli enzimi che possono legare il DNA e, tra questi, sono particolarmente importanti le polimerasi che copiano le sequenze nella trascrizione e nella replicazione del DNA.
Proteine che legano il DNA
Le proteine strutturali che legano il DNA sono esempi delle interazioni aspecifiche tra DNA e proteine. All'interno dei cromosomi, il DNA è associato a complessi di natura proteica, che si organizzano tra loro a formare una struttura compatta chiamata cromatina . Negli eucarioti, questa struttura presuppone il legame del DNA a piccoli complessi proteici basici chiamati istoni ; nei procarioti, invece, sono coinvolti diversi tipi di differenti proteine. [93] [94] Gli istoni formano un complesso a forma di disco chiamato nucleosoma , che instaura interazioni di tipo ionico (tra i residui basici degli istoni e lo scheletro fosforico acido del DNA) con circa duecento paia di basi di DNA, che si avvolgono intorno al disco formando due giri completi, indipendentemente dalla sequenza che li caratterizza. [95] Questi residui basici possono subire metilazioni , fosforilazioni e acetilazioni : [96] tali modificazioni chimiche alterano l'interazione tra gli istoni e il DNA, rendendolo così più o meno accessibile ai fattori di trascrizione e modulando la velocità della trascrizione stessa. [97]
Altre DNA-binding proteins (DNAbp) di tipo aspecifico, anch'esse presenti nella cromatina , sono le high-mobility group proteins , che legano preferenzialmente il DNA ripiegato o distorto. [98] Queste proteine hanno un ruolo fondamentale nel ripiegamento delle file di nucleosomi e nel loro impacchettamento all'interno di strutture cromatiniche più complesse. [99]
Un ulteriore gruppo di DNAbp sono le single-strand-binding proteins (SSBP), che si legano esclusivamente a una molecola di DNA a singolo filamento. Nell'uomo, la RPA ( replication protein A ) è il membro meglio caratterizzato di questa famiglia ed è essenziale per la maggior parte dei processi che richiedono una separazione della doppia elica, tra cui la replicazione del DNA, la sua ricombinazione e la sua riparazione . [100] Queste DNAbp sono in grado di stabilizzare la forma a singolo filamento, impedendo che la molecola si ripieghi a formare stem loops o venga degradata dall'azione delle nucleasi .

A differenza di quelle finora presentate, esistono anche numerose proteine che legano specificamente determinate sequenze di DNA. Quelle maggiormente studiate sono i fattori di trascrizione (TF), proteine in grado di regolare la trascrizione. Ognuna di esse si lega ad una o più sequenze specifiche, poste solitamente nei pressi del promotore di un gene, attivando o inibendo la trascrizione del gene stesso. Tale processo viene svolto attraverso due tipi di meccanismo: anzitutto, i TF sono in grado di legare, direttamente o attraverso proteine adattatrici, la RNA polimerasi responsabile della trascrizione, localizzandola presso il sito di inizio della trascrizione e favorendone dunque l'avvio; [102] un'altra modalità consiste nel legame tra i TF ed enzimi in grado di modulare metilazioni ed acetilazioni degli istoni presenti presso il promotore, modificando dunque l'accessibilità di quella regione alla polimerasi. [103]
Dal momento che un TF può avere numerose sequenze bersaglio, cambiamenti di attività di un TF possono avere effetti sull'espressione di migliaia di geni. [104] Di conseguenza, queste proteine sono spesso i bersagli finali delle cascate di trasduzione del segnale , che mediano le risposte cellulari agli stimoli interni ed esterni alla cellula. La specificità dei TF per il DNA è legato ai contatti multipli che si instaurano tra la proteina ed il solco maggiore, dove le basi azotate sono maggiormente accessibili. [33]
Enzimi che modificano il DNA
Nucleasi e ligasi
Le nucleasi sono enzimi in grado di tagliare filamenti di DNA, dal momento che catalizzano l' idrolisi del legame fosfodiesterico . Le nucleasi che idrolizzano il DNA partendo dai nucleotidi situati alle estremità dei filamenti sono definite esonucleasi . Sono endonucleasi , invece, quelle che tagliano direttamente all'interno del filamento. Le nucleasi più utilizzate in biologia molecolare , dette enzimi di restrizione , tagliano il DNA in corrispondenza di specifiche sequenze. L'enzima EcoRV , ad esempio, riconosce la sequenza di sei basi 5′-GAT|ATC-3′ ed effettua il taglio presso la linea verticale. In natura, questo enzima protegge i batteri dalle infezioni fagiche , digerendo il DNA del fago quando esso fa il suo ingresso nella cellula batterica. [106] Generalmente, le nucleasi di restrizione riconoscono particolari sequenze nucleotidiche palindromiche , note come siti di restrizione , nelle quali la stessa sequenza si ripete in direzioni opposte sulle due eliche complementari, e quindi producono dei tagli su entrambi i filamenti. Tali enzimi sono utilizzati ampiamente nelle tecniche che prevedono il subclonaggio di DNA all'interno di vettori . Si crede che le sequenze palindromiche possano essere, oltre a sti di restrizione, anche segnali di riconoscimento per alcune proteine di regolazione oppure segnalare il sito di avvio della replicazione del DNA.
Le DNA ligasi sono enzimi in grado di riunire filamenti di DNA precedentemente tagliati o spezzati, utilizzando energia chimica proveniente da ATP o da NAD . [107] Le ligasi sono particolarmente importanti nella replicazione del filamento lento , dal momento che esse riuniscono i frammenti di Okazaki in un filamento unico. Esse rivestono un ruolo importante anche nella riparazione del DNA e nella ricombinazione genetica . [107]
Topoisomerasi ed elicasi
Le topoisomerasi sono enzimi che presentano sia un'attività nucleasica che una ligasica . Queste proteine sono in grado di modificare le proprietà topologiche del DNA. Alcune di esse svolgono tale funzione tagliando l'elica di DNA e permettendole di ruotare, riducendo il suo grado di superavvolgimento, per poi procedere alla ligazione delle due estremità. [48] Altre topoisomerasi sono invece in grado di tagliare l'elica e far passare attraverso il sito di rottura una seconda elica, prima di ligare il filamento spezzato. [108] Le topoisomerasi sono necessarie per molti processi che coinvolgono il DNA, come ad esempio la replicazione del DNA e la trascrizione. [49]
Le elicasi sono proteine in grado di utilizzare l' energia chimica presente nei nucleosidi trifosfato , soprattutto ATP , per rompere i legami idrogeno che si instaurano tra le basi azotate, permettendo l'apertura della doppia elica di DNA in singoli filamenti. [109] Questi enzimi sono essenziali per la maggior parte dei processi biologici che coinvolgono enzimi che richiedono un diretto contatto con le basi del DNA.
Polimerasi
Le polimerasi sono enzimi che sintetizzano catene polinucleotidiche a partire dal nucleosidi trifosfato . Esse funzionano aggiungendo nucleotidi al 3′- OH del precedente nucleotide presente sul filamento. Come conseguenza di ciò, tutte le polimerasi lavorano in direzione 5′ - 3′ . [110] Nel sito attivo di questi enzimi, il nucleoside trifosfato si appaia ad un nucleotide presente su un filamento usato come stampo: ciò permette alle polimerasi di sintetizzare in modo accurato filamenti fedelmente complementari agli stampi. Le polimerasi sono classificate sulla base del tipo di stampo che utilizzano.
La replicazione del DNA richiede una DNA polimerasi DNA-dipendente , in grado cioè di realizzare una perfetta copia di una sequenza di DNA. L'accuratezza è fondamentale in questo processo, motivo per cui molte di queste polimerasi presentano anche un'attività di proofreading (dall' inglese , correzione di bozze ). Esse sono infatti in grado di rilevare un errore di appaiamento (o mismatch ) tra basi azotate e attivare un'azione 3' o 5' esonucleasica per rimuovere la base scorretta. [111] Nella maggior parte degli organismi, le DNA polimerasi funzionano all'interno di un più ampio complesso proteico definito replisoma , che consiste anche di numerose subunità accessorie come ad esempio le elicasi . [112]
Le DNA polimerasi RNA-dipendenti sono una classe di polimerasi specializzate nella sintesi di una copia di DNA, usando come stampo un frammento di RNA. Tra di esse figurano la trascrittasi inversa , un enzima virale coinvolto nell'infezione dei retrovirus , e la telomerasi , necessaria per la replicazione dei telomeri . [62] [113] La telomerasi è una polimerasi inusuale, dal momento che contiene una sequenza stampo di RNA all'interno della propria struttura.
La trascrizione è invece svolta da RNA polimerasi DNA-dipendenti , che legano il DNA presso il promotore di un gene e separano i due filamenti. Successivamente, l'enzima genera una molecola di mRNA fino al raggiungimento del terminatore , dove si interrompe la trascrizione e l'enzima si distacca dal DNA. Come avviene per le DNA polimerasi DNA-dipendenti, anche queste polimerasi operano all'interno di un ampio complesso proteico, composto di molecole accessorie e regolatorie. [114]
Ricombinazione genetica
 |
 |
Un filamento di DNA solitamente non interagisce con altri segmenti di DNA e, nelle cellule umane, i differenti cromosomi occupano addirittura regioni separate del nucleo ( territori cromosomici ). [116] Tale separazione fisica è fondamentale per permettere al DNA di essere un archivio stabile e sicuro dell'informazione genetica. L'interazione tra diversi segmenti di DNA è invece possibile e frequente attraverso il fenomeno del crossing-over , che permette la ricombinazione genetica attraverso la rottura di due eliche, lo scambio di segmenti tra di esse ed il ricongiungimento finale.
La ricombinazione permette ai cromosomi di scambiare informazioni genetiche e produrre nuove combinazioni di geni, con il risultato di aumentare l'efficienza della selezione naturale e di facilitare l'evoluzione di nuove proteine. [117] La ricombinazione genetica può anche essere coinvolta nella riparazione del DNA, in particolare come risposta cellulare in seguito a rotture a doppio filamento. [118]
La principale forma di crossing-over cromosomico è la ricombinazione omologa , nella quale i due cromosomi coinvolti condividono sequenze molto simili. Le ricombinazioni non omologhe, invece, possono essere dannose per la cellula, perché in grado di produrre traslocazioni cromosomiche e anomalie genetiche. La reazione di ricombinazione è catalizzata da enzimi noti come ricombinasi . [119] Il primo passaggio del processo di ricombinazione consiste nella rottura a singolo filamento provocata da un'endonucleasi o da un danno al DNA. [120] Una serie di passaggi successivi, in parte catalizzati dalla ricombinasi, porta all'unione tra due eliche attraverso la formazione di una giunzione di Holliday , nella quale un segmento a singolo filamento di ogni elica è appaiato al filamento complementare presente sull'altra elica. La reazione di ricombinazione è quindi interrotta dalla rottura della giunzione e dalla re-ligazione del DNA così ottenuto. [121] L'esistenza della giunzione di Holliday è stata dimostrata da fotografie al microscopio elettronico di molecole di DNA in ricombinazione.
Evoluzione del metabolismo del DNA
Il DNA contiene l'informazione genetica che permette a tutti gli organismi viventi (esclusi i virus, la cui ammissibilità tra i viventi è tuttavia ampiamente dibattuta) di funzionare, crescere e riprodursi. In ogni caso, non è stato ancora chiarito in quale momento della storia della vita il DNA abbia assunto tale ruolo fondamentale. È generalmente accettato dalla comunità scientifica, infatti, l' ipotesi che il DNA non sia stato il primo acido nucleico ad essere utilizzato dai viventi: tale ruolo spetterebbe infatti all'RNA. [122] [123] L'RNA potrebbe aver giocato un ruolo centrale del metabolismo cellulare ancestrale, dal momento che può avere sia un ruolo nella conservazione dell'informazione genetica, sia uno catalitico (ad esempio attraverso i ribozimi ), sia uno strutturale (all'interno dei ribosomi ). [124] Il mondo a RNA , basato su un unico tipo di molecola avente funzioni genetiche, catalitiche e strutturali, potrebbe avere avuto un'influenza sullo sviluppo dell'attuale codice genetico, basato proprio su quattro nucleotidi. Tale numero potrebbe essere un compromesso tra la necessità da una parte di ridurre la quantità di basi possibili, per migliorare l'accuratezza della replicazione, e dall'altra di aumentarla, per incrementare l'efficacia catalitica dei ribozimi. [125]
Non si conoscono fossili di DNA risalenti all'origine della vita (dopo il mondo a RNA) per poterne studiare l'evoluzione molecolare, dal momento che è impossibile recuperare DNA dai fossili. Ciò è dovuto al fatto che il DNA può sopravvivere nell'ambiente per meno di un milione di anni e, se in soluzione, si degrada rapidamente in piccoli frammenti. [126] Sebbene ci siano stati diversi annunci di scoperte di DNA antichissimo, tra cui quella relativa all'isolamento di un batterio vivo da un cristallo di sale risalente a 250 milioni di anni fa, [127] queste affermazioni sono controverse. [128] [129]
Utilizzi del DNA
Ingegneria genetica
La moderna biologia e biochimica fa un uso intensivo del DNA. Con il termine di DNA ricombinante ci si riferisce a segmenti di DNA realizzati e assemblati artificialmente. Essi possono essere inseriti all'interno di organismi viventi sotto forma di plasmidi o mediante altri tipi di vettori . [130] Gli organismi così prodotti sono detti geneticamente modificati e possono essere utilizzati per la produzione di proteine ricombinanti, necessarie per la ricerca biomedica, [131] o per le coltivazioni agricole . [132] [133]
Medicina forense
La medicina forense si serve del DNA, generalmente isolato dal sangue , dalla pelle , dalla saliva , dai capelli e da altri tessuti e fluidi biologici, per identificare i responsabili di atti criminosi, come delitti o violenze. Il processo utilizzato è il fingerprinting genetico : tale tecnica consiste nel comparare la lunghezza delle sezioni variabili del DNA ripetitivo , come le short tandem repeats ed i minisatelliti , che possono risultare molto diverse tra un individuo e l'altro. La comparazione tra due campioni di DNA in esame, non si basa perciò sull'analisi di tutta la sequenza desossiribonucleotidica, ma solo su tali sezioni. Infatti, due individui non legati da rapporti di parentela hanno in comune ben il 99,9% di sequenza di DNA. Tale metodo è solitamente molto affidabile, [134] anche se a volte l'identificazione dei criminali può risultare complicata qualora la scena sia contaminata dal DNA di diverse persone. [135] Questo metodo, sviluppato nel 1984 dal genetista britannico Sir Alec Jeffreys , [136] fu usato per la prima volta nel 1988 per incriminare Colin Pitchfork. Nella pratica attuale, spesso i sospettati sono invitati a fornire un campione di DNA per il confronto con eventuali reperti biologici presenti sulla scena del delitto. Il fingerprinting genetico può essere utilizzato anche per identificare le vittime di incidenti di massa.
L'acquisizione del DNA senza consenso viene indicato con il termine di Gene theft .
Bioinformatica
La bioinformatica è una branca della biologia che comprende la manipolazione, la ricerca ed il data mining dei dati relativi a sequenze di DNA. Lo sviluppo di tecniche utili ad immagazzinare e ricercare sequenze di DNA, infatti, ha condotto ad ampi sviluppi dell'informatica applicata alla biologia molecolare, specialmente per quanto riguarda gli algoritmi di ricerca di stringhe e l' apprendimento automatico . [137] Gli algoritmi di ricerca (o appaiamento) di stringhe, in grado di individuare la presenza di una sequenza di lettere all'interno di sequenze molto più ampie, furono inizialmente sviluppati per la ricerca di specifiche sequenze nucleotidiche. [138]
Esistono da molto tempo, ovviamente, semplici algoritmi in grado di affrontare questi problemi (quelli presenti, ad esempio, negli editor di testo ), ma l'analisi del DNA, che si presenta come composto di sole quattro lettere, richiede programmi più elaborati. Il problema immediatamente correlato dell' allineamento di sequenze si pone come obiettivo quello di identificare le sequenze omologhe ed individuare le specifiche mutazioni che le rendono differenti. Queste tecniche, in particolare l' allineamento di sequenze multiple , sono utilizzate per studiare le relazioni filogenetiche e la funzione delle proteine. [139] Esistono anche algoritmi di ricerca genetica.
La grande quantità di dati ottenuta da progetti come il progetto genoma umano è infatti di difficile utilizzo senza una prima analisi che permetta di localizzare i geni e le regioni regolatorie sui cromosomi. Tali algoritmi, dunque, sono in grado di riconoscere regioni putative di presenza di geni codificanti RNA o proteine. [140]
DNA in informatica e nanotecnologie

Il DNA è stato utilizzato in informatica per la prima volta per risolvere un semplice problema di cammino hamiltoniano , un problema NP-completo . [142] Il calcolo attraverso il DNA è più vantaggioso rispetto a quello classico per via elettronica sia dal punto di vista dell'energia consumata, sia da quello dello spazio utilizzato: strutture di questo genere sono infatti in grado di svolgere calcoli in modalità parallele che permettono di risolvere agevolmente numerosi altri problemi quali la simulazione di macchine astratte , il problema di soddisfacibilità booleana e la versione bounded del problema del commesso viaggiatore . [143] Grazie alla sua compattezza, il DNA presenta anche un ruolo (almeno teorico) nel campo della crittografia , nella quale permetterebbe in particolare la costituzione e l'utilizzo efficiente di cifrari di Vernam sicuri. [144]
Il DNA è utilizzato anche nel campo delle nanotecnologie poiché presenta proprietà di riconoscimento molecolare che lo rendono in grado di auto-assemblarsi in strutture complesse di tipo bidimensionale o poliedrico . Tali assemblati sono utilizzati con funzioni essenzialmente strutturali e non come vettori di informazione biologica. [145]
Storia e antropologia
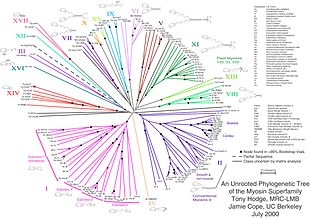
Dal momento che il DNA è sottoposto nel tempo a mutazioni che vengono ereditate, esso contiene informazioni preziose che possono essere utilizzate dai genetisti per studiare l'evoluzione degli organismi e la loro filogenesi . [147] Sulla base delle diverse mutazioni presenti in geni estremamente conservati tra gli organismi (oppure, tramite algoritmi comparativi bioinformatici più avanzati, confrontando direttamente interi genomi) i genetisti sono in grado di ricostruire alberi filogenetici in grado di descrivere l'evoluzione di diverse specie anche molto diverse tra loro. [148] [149] Studiando le mutazioni accumulatesi nel tempo, è anche possibile ricostruire alberi che descrivano l'evoluzione all'interno di famiglie di proteine.
Comparando le sequenze di DNA all'interno di una stessa specie, inoltre, è possibile studiare la storia genetica di particolari popolazioni . Ciò presenta una notevole rilevanza sia per analisi ecologiche sia per studi antropologici : il DNA è stato usato, ad esempio, per ricostruire la vicenda delle dieci tribù perdute d'Israele . [150]
Note
- ^ ADN , in Treccani.it – Vocabolario Treccani on line , Istituto dell'Enciclopedia Italiana. URL consultato il 27 aprile 2017 .
- ^ Lemma " ADN " in Tullio De Mauro , Dizionario italiano .
- ^ Cfr. " DNA o ADN " nell'enciclopedia Sapere .
- ^ Esistono numerosi virus privi di DNA, ma la loro classificazione come organismi viventi è tuttora dibattuta
- ^ Dahm R, Friedrich Miescher and the discovery of DNA , in Dev Biol , vol. 278, n. 2, 2005, pp. 274–88, PMID 15680349 .
- ^ Levene P,, The structure of yeast nucleic acid , in J Biol Chem , vol. 40, n. 2, 1919, pp. 415–24.
- ^ Astbury W,, Nucleic acid , in Symp. SOC. Exp. Bbl , vol. 1, n. 66, 1947.
- ^ Erwin Schrödinger - What is Life? The Physical Aspect of the Living Cell , Cambridge University Press, Cambridge 1944
- ^ Lorenz MG, Wackernagel W, Bacterial gene transfer by natural genetic transformation in the environment , in Microbiol. Rev. , vol. 58, n. 3, 1994, pp. 563–602, PMID 7968924 .
- ^ Avery O, MacLeod C, McCarty M, Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III , in J Exp Med , vol. 79, n. 2, 1944, pp. 137–158.
- ^ Hershey A, Chase M, Independent functions of viral protein and nucleic acid in growth of bacteriophage ( PDF ), in J Gen Physiol , vol. 36, n. 1, 1952, pp. 39–56, PMID 12981234 .
- ^ a b Watson JD and Crick FHC "A Structure for Deoxyribose Nucleic Acid". (PDF) Nature 171, 737–738 (1953). Ultimo accesso: 13 febbraio 2007.
- ^ a b Watson J, Crick F,Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid ( PDF ), in Nature , vol. 171, n. 4356, 1953, pp. 737–8, PMID 13054692 .
- ^ Nature Archives Double Helix of DNA: 50 Years
- ^ Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling RGNature 171, 740–741 (1953) Nature Archives Full Text (PDF)
- ^ Original X-ray diffraction image
- ^ Molecular Structure of Deoxypentose Nucleic Acids. Wilkins MHF, AR Stokes AR & Wilson, HR Nature 171, 738–740 (1953) Nature Archives (PDF)
- ^ The Nobel Prize in Physiology or Medicine 1962 Nobelprize.org. Ultimo accesso: 22 dicembre 2006
- ^ Crick, FHC On degenerate templates and the adaptor hypothesis (PDF). Archiviato il 1º ottobre 2008 in Internet Archive . genome.wellcome.ac.uk (Lecture, 1955). Ultimo accesso: 22 dicembre 2006
- ^ Meselson M, Stahl F, The replication of DNA in Escherichia coli , in Proc Natl Acad Sci USA , vol. 44, n. 7, 1958, pp. 671–82, PMID 16590258 .
- ^ The Nobel Prize in Physiology or Medicine 1968 Nobelprize.org. Ultimo accesso: 22 dicembre 2006
- ^ a b Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters, Molecular Biology of the Cell; Fourth Edition , New York and London, Garland Science, 2002, ISBN 0-8153-3218-1 .
- ^ Butler, John M. (2001) Forensic DNA Typing "Elsevier". pp. 14–15. ISBN 978-0-12-147951-0 .
- ^ Mandelkern M, Elias J, Eden D, Crothers D, The dimensions of DNA in solution , in J Mol Biol , vol. 152, n. 1, 1981, pp. 153–61, PMID 7338906 .
- ^ Gregory S, et al. , The DNA sequence and biological annotation of human chromosome 1 , in Nature , vol. 441, n. 7091, 2006, pp. 315–21, PMID 16710414 .
- ^ a b c Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. WH Freeman and Company ISBN 0-7167-4955-6
- ^ Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN). Ultimo accesso: 3 gennaio 2006
- ^ a b Ghosh A, Bansal M, A glossary of DNA structures from A to Z , in Acta Crystallogr D Biol Crystallogr , vol. 59, Pt 4, 2003, pp. 620–6, PMID 12657780 .
- ^ ( EN ) IUPAC Gold Book, "deoxyribonucleic acids (DNA)"
- ^ Takahashi I, Marmur J., Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis , in Nature , vol. 197, 1963, pp. 794–5, PMID 13980287 .
- ^ Agris P, Decoding the genome: a modified view , in Nucleic Acids Res , vol. 32, n. 1, 2004, pp. 223–38, PMID 14715921 .
- ^ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R, Crystal structure analysis of a complete turn of B-DNA , in Nature , vol. 287, n. 5784, 1980, pp. 755–8, PMID 7432492 .
- ^ a b Pabo C, Sauer R, Protein-DNA recognition , in Annu Rev Biochem , vol. 53, 1984, pp. 293–321, PMID 6236744 .
- ^ Tra le eccezioni a questa regola, si ricordano le proteine con dominio di legame al DNA di tipo HMG-box , quale HMGB1 .
- ^ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H, Mechanical stability of single DNA molecules , in Biophys J , vol. 78, n. 4, 2000, pp. 1997–2007, PMID 10733978 .
- ^ Chalikian T, Völker J, Plum G, Breslauer K, A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques , in Proc Natl Acad Sci USA , vol. 96, n. 14, 1999, pp. 7853–8, PMID 10393911 .
- ^ deHaseth P, Helmann J, Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA , in Mol Microbiol , vol. 16, n. 5, 1995, pp. 817–24, PMID 7476180 .
- ^ Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J, Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern , in Biochemistry , vol. 43, n. 51, 2004, pp. 15996–6010, PMID 15609994 .
- ^ Ponnuswamy P, Gromiha M, On the conformational stability of oligonucleotide duplexes and tRNA molecules , in J Theor Biol , vol. 169, n. 4, 1994, pp. 419–32, PMID 7526075 .
- ^ Hüttenhofer A, Schattner P, Polacek N, Non-coding RNAs: hope or hype? , in Trends Genet , vol. 21, n. 5, 2005, pp. 289–97, PMID 15851066 .
- ^ Munroe S, Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns , in J Cell Biochem , vol. 93, n. 4, 2004, pp. 664–71, PMID 15389973 .
- ^ Makalowska I, Lin C, Makalowski W, Overlapping genes in vertebrate genomes , in Comput Biol Chem , vol. 29, n. 1, 2005, pp. 1–12, PMID 15680581 .
- ^ Johnson Z, Chisholm S, Properties of overlapping genes are conserved across microbial genomes , in Genome Res , vol. 14, n. 11, 2004, pp. 2268–72, PMID 15520290 .
- ^ Lamb R, Horvath C, Diversity of coding strategies in influenza viruses , in Trends Genet , vol. 7, n. 8, 1991, pp. 261–6, PMID 1771674 .
- ^ Davies J, Stanley J, Geminivirus genes and vectors , in Trends Genet , vol. 5, n. 3, 1989, pp. 77–81, PMID 2660364 .
- ^ Berns K, Parvovirus replication , in Microbiol Rev , vol. 54, n. 3, 1990, pp. 316–29, PMID 2215424 .
- ^ Benham C, Mielke S, DNA mechanics , in Annu Rev Biomed Eng , vol. 7, 2005, pp. 21–53, PMID 16004565 .
- ^ a b Champoux J, DNA topoisomerases: structure, function, and mechanism , in Annu Rev Biochem , vol. 70, 2001, pp. 369–413, PMID 11395412 .
- ^ a b Wang J, Cellular roles of DNA topoisomerases: a molecular perspective , in Nat Rev Mol Cell Biol , vol. 3, n. 6, 2002, pp. 430–40, PMID 12042765 .
- ^ a b Hayashi G, Hagihara M, Nakatani K, Application of L-DNA as a molecular tag , in Nucleic Acids Symp Ser (Oxf) , vol. 49, 2005, pp. 261–262, PMID 17150733 .
- ^ Vargason JM, Eichman BF, Ho PS, The extended and eccentric E-DNA structure induced by cytosine methylation or bromination , in Nature Structural Biology , vol. 7, 2000, pp. 758–761, PMID 10966645 .
- ^ Wang G, Vasquez KM, Non-B DNA structure-induced genetic instability , in Mutat Res , vol. 598, 1–2, 2006, pp. 103–119, PMID 16516932 .
- ^ Allemand, et al, Stretched and overwound DNA forms a Pauling-like structure with exposed bases , in PNAS , vol. 24, 1998, pp. 14152-14157, PMID 9826669 .
- ^ Palecek E, Local supercoil-stabilized DNA structures , in Critical Reviews in Biochemistry and Molecular Biology , vol. 26, n. 2, 1991, pp. 151–226, PMID 1914495 .
- ^ Basu H, Feuerstein B, Zarling D, Shafer R, Marton L, Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies , in J Biomol Struct Dyn , vol. 6, n. 2, 1988, pp. 299–309, PMID 2482766 .
- ^ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL, Polymorphism of DNA double helices , in J. Mol. Biol. , vol. 143, n. 1, 1980, pp. 49–72, PMID 7441761 .
- ^ Wahl M, Sundaralingam M, Crystal structures of A-DNA duplexes , in Biopolymers , vol. 44, n. 1, 1997, pp. 45–63, PMID 9097733 .
- ^ Lu XJ, Shakked Z, Olson WK, A-form conformational motifs in ligand-bound DNA structures , in J. Mol. Biol. , vol. 300, n. 4, 2000, pp. 819-40, PMID 10891271 .
- ^ Rothenburg S, Koch-Nolte F, Haag F, DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles , in Immunol Rev , vol. 184, pp. 286–98, PMID 12086319 .
- ^ Oh D, Kim Y, Rich A, Z-DNA-binding proteins can act as potent effectors of gene expression in vivo , in Proc. Natl. Acad. Sci. USA , vol. 99, n. 26, 2002, pp. 16666-71, PMID 12486233 .
- ^ Immagine realizzata da NDB UD0017 Archiviato il 7 giugno 2013 in Internet Archive .
- ^ a b Greider C, Blackburn E, Identification of a specific telomere terminal transferase activity in Tetrahymena extracts , in Cell , vol. 43, 2 Pt 1, 1985, pp. 405–13, PMID 3907856 .
- ^ Wright W, Tesmer V, Huffman K, Levene S, Shay J, Normal human chromosomes have long G-rich telomeric overhangs at one end , in Genes Dev , vol. 11, n. 21, 1997, pp. 2801–9, PMID 9353250 .
- ^ a b Burge S, Parkinson G, Hazel P, Todd A, Neidle S, Quadruplex DNA: sequence, topology and structure , in Nucleic Acids Res , vol. 34, n. 19, 2006, pp. 5402–15, PMID 17012276 .
- ^ Parkinson G, Lee M, Neidle S, Crystal structure of parallel quadruplexes from human telomeric DNA , in Nature , vol. 417, n. 6891, 2002, pp. 876–80, PMID 12050675 .
- ^ Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T, Mammalian telomeres end in a large duplex loop , in Cell , vol. 97, n. 4, 1999, pp. 503–14, PMID 10338214 .
- ^ Klose R, Bird A, Genomic DNA methylation: the mark and its mediators , in Trends Biochem Sci , vol. 31, n. 2, 2006, pp. 89–97, PMID 16403636 .
- ^ Bird A, DNA methylation patterns and epigenetic memory , in Genes Dev , vol. 16, n. 1, 2002, pp. 6–21, PMID 11782440 .
- ^ Walsh C, Xu G, Cytosine methylation and DNA repair , in Curr Top Microbiol Immunol , vol. 301, pp. 283–315, PMID 16570853 .
- ^ Ratel D, Ravanat J, Berger F, Wion D, N6-methyladenine: the other methylated base of DNA , in Bioessays , vol. 28, n. 3, 2006, pp. 309–15, PMID 16479578 .
- ^ Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P, beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei , in Cell , vol. 75, n. 6, 1993, pp. 1129–36, PMID 8261512 .
- ^ Immagine realizzata a partire dalla entry PDB 1JDG Archiviato il 22 settembre 2008 in Internet Archive .
- ^ Douki T, Reynaud-Angelin A, Cadet J, Sage E, Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation , in Biochemistry , vol. 42, n. 30, 2003, pp. 9221–6, PMID 12885257 .
- ^ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S, Hydroxyl radicals and DNA base damage , in Mutat Res , vol. 424, 1–2, 1999, pp. 9–21, PMID 10064846 .
- ^ Shigenaga M, Gimeno C, Ames B, Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker of in vivo oxidative DNA damage , in Proc Natl Acad Sci USA , vol. 86, n. 24, 1989, pp. 9697–701, PMID 2602371 .
- ^ Cathcart R, Schwiers E, Saul R, Ames B, Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage ( PDF ), in Proc Natl Acad Sci USA , vol. 81, n. 18, 1984, pp. 5633–7, PMID 6592579 .
- ^ Valerie K, Povirk L, Regulation and mechanisms of mammalian double-strand break repair , in Oncogene , vol. 22, n. 37, 2003, pp. 5792–812, PMID 12947387 .
- ^ Ferguson L, Denny W, The genetic toxicology of acridines , in Mutat Res , vol. 258, n. 2, 1991, pp. 123–60, PMID 1881402 .
- ^ Jeffrey A, DNA modification by chemical carcinogens , in Pharmacol Ther , vol. 28, n. 2, 1985, pp. 237–72, PMID 3936066 .
- ^ Stephens T, Bunde C, Fillmore B, Mechanism of action in thalidomide teratogenesis , in Biochem Pharmacol , vol. 59, n. 12, 2000, pp. 1489–99, PMID 10799645 .
- ^ Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A, Intercalators as anticancer drugs , in Curr Pharm Des , vol. 7, n. 17, 2001, pp. 1745–80, PMID 11562309 .
- ^ Venter J, et al. , The sequence of the human genome , in Science , vol. 291, n. 5507, 2001, pp. 1304–51, PMID 11181995 .
- ^ Thanbichler M, Wang S, Shapiro L, The bacterial nucleoid: a highly organized and dynamic structure , in J Cell Biochem , vol. 96, n. 3, 2005, pp. 506–21, PMID 15988757 .
- ^ Wolfsberg T, McEntyre J, Schuler G, Guide to the draft human genome , in Nature , vol. 409, n. 6822, 2001, pp. 824–6, PMID 11236998 .
- ^ Gregory T, The C-value enigma in plants and animals: a review of parallels and an appeal for partnership , in Ann Bot (Lond) , vol. 95, n. 1, 2005, pp. 133–46, PMID 15596463 .
- ^ The ENCODE Project Consortium, Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project , in Nature , vol. 447, n. 7146, 2007, pp. 799-816, DOI : 10.1038/nature05874 .
- ^ Realizzata a partire dall'entry PDB 1MSW
- ^ Pidoux A, Allshire R, The role of heterochromatin in centromere function , in Philos Trans R Soc Lond B Biol Sci , vol. 360, n. 1455, 2005, pp. 569–79, PMID 15905142 .
- ^ Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M, Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22 , in Genome Res , vol. 12, n. 2, 2002, pp. 272–80, PMID 11827946 .
- ^ Harrison P, Gerstein M, Studying genomes through the aeons: protein families, pseudogenes and proteome evolution , in J Mol Biol , vol. 318, n. 5, 2002, pp. 1155–74, PMID 12083509 .
- ^ Animazione esplicativa dell'esperimento di Kornberg del 1958 )
- ^ Albà M, Replicative DNA polymerases , in Genome Biol , vol. 2, n. 1, 2001, pp. REVIEWS3002, PMID 11178285 .
- ^ Sandman K, Pereira S, Reeve J, Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome , in Cell Mol Life Sci , vol. 54, n. 12, 1998, pp. 1350–64, PMID 9893710 .
- ^ Dame RT, The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin , in Mol. Microbiol. , vol. 56, n. 4, 2005, pp. 858-70, PMID 15853876 .
- ^ Luger K, Mäder A, Richmond R, Sargent D, Richmond T, Crystal structure of the nucleosome core particle at 2.8 A resolution , in Nature , vol. 389, n. 6648, 1997, pp. 251–60, PMID 9305837 .
- ^ Jenuwein T, Allis C, Translating the histone code , in Science , vol. 293, n. 5532, 2001, pp. 1074–80, PMID 11498575 .
- ^ Ito T, Nucleosome assembly and remodelling , in Curr Top Microbiol Immunol , vol. 274, pp. 1–22, PMID 12596902 .
- ^ Thomas J, HMG1 and 2: architectural DNA-binding proteins , in Biochem Soc Trans , vol. 29, Pt 4, 2001, pp. 395–401, PMID 11497996 .
- ^ Grosschedl R, Giese K, Pagel J, HMG domain proteins: architectural elements in the assembly of nucleoprotein structures , in Trends Genet , vol. 10, n. 3, 1994, pp. 94–100, PMID 8178371 .
- ^ Iftode C, Daniely Y, Borowiec J, Replication protein A (RPA): the eukaryotic SSB , in Crit Rev Biochem Mol Biol , vol. 34, n. 3, 1999, pp. 141–80, PMID 10473346 .
- ^ Realizzato a partire dalla entry PDB 1LMB
- ^ Myers L, Kornberg R, Mediator of transcriptional regulation , in Annu Rev Biochem , vol. 69, 2000, pp. 729–49, PMID 10966474 .
- ^ Spiegelman B, Heinrich R, Biological control through regulated transcriptional coactivators , in Cell , vol. 119, n. 2, 2004, pp. 157-67, PMID 15479634 .
- ^ Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B, A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells , in Proc Natl Acad Sci USA , vol. 100, n. 14, 2003, pp. 8164–9, PMID 12808131 .
- ^ Immagine realizzata a partire dalla entry PDB 1RVA
- ^ Bickle T, Krüger D, Biology of DNA restriction , in Microbiol Rev , vol. 57, n. 2, 1993, pp. 434–50, PMID 8336674 .
- ^ a b Doherty A, Suh S, Structural and mechanistic conservation in DNA ligases. , in Nucleic Acids Res , vol. 28, n. 21, 2000, pp. 4051–8, PMID 11058099 .
- ^ Schoeffler A, Berger J, Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism , in Biochem Soc Trans , vol. 33, Pt 6, 2005, pp. 1465–70, PMID 16246147 .
- ^ Tuteja N, Tuteja R, Unraveling DNA helicases. Motif, structure, mechanism and function [ collegamento interrotto ] , in Eur J Biochem , vol. 271, n. 10, 2004, pp. 1849–63, DOI : 10.1111/j.1432-1033.2004.04094.x , PMID 15128295 .
- ^ Joyce C, Steitz T, Polymerase structures and function: variations on a theme? , in J Bacteriol , vol. 177, n. 22, 1995, pp. 6321–9, PMID 7592405 .
- ^ Hubscher U, Maga G, Spadari S, Eukaryotic DNA polymerases , in Annu Rev Biochem , vol. 71, 2002, pp. 133–63, PMID 12045093 .
- ^ Johnson A, O'Donnell M, Cellular DNA replicases: components and dynamics at the replication fork , in Annu Rev Biochem , vol. 74, 2005, pp. 283–315, PMID 15952889 .
- ^ Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S, The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention , in FASEB J , vol. 8, n. 8, 1994, pp. 497–503, PMID 7514143 .
- ^ Martinez E, Multi-protein complexes in eukaryotic gene transcription , in Plant Mol Biol , vol. 50, n. 6, 2002, pp. 925–47, PMID 12516863 .
- ^ Immagine realizzata a partire dalla entry PDB 1M6G
- ^ Cremer T, Cremer C, Chromosome territories, nuclear architecture and gene regulation in mammalian cells , in Nat Rev Genet , vol. 2, n. 4, 2001, pp. 292–301, PMID 11283701 .
- ^ Pál C, Papp B, Lercher M, An integrated view of protein evolution , in Nat Rev Genet , vol. 7, n. 5, 2006, pp. 337–48, PMID 16619049 .
- ^ O'Driscoll M, Jeggo P, The role of double-strand break repair - insights from human genetics , in Nat Rev Genet , vol. 7, n. 1, 2006, pp. 45–54, PMID 16369571 .
- ^ Vispé S, Defais M, Mammalian Rad51 protein: a RecA homologue with pleiotropic functions , in Biochimie , vol. 79, n. 9-10, 1997, pp. 587-92, PMID 9466696 .
- ^ Neale MJ, Keeney S, Clarifying the mechanics of DNA strand exchange in meiotic recombination , in Nature , vol. 442, n. 7099, 2006, pp. 153-8, PMID 16838012 .
- ^ Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D, The RuvABC resolvasome , in Eur J Biochem , vol. 269, n. 22, 2002, pp. 5492–501, PMID 12423347 .
- ^ Joyce G, The antiquity of RNA-based evolution , in Nature , vol. 418, n. 6894, 2002, pp. 214–21, PMID 12110897 .
- ^ Orgel L, Prebiotic chemistry and the origin of the RNA world ( PDF ), in Crit Rev Biochem Mol Biol , vol. 39, n. 2, pp. 99–123, PMID 15217990 .
- ^ Davenport R, Ribozymes. Making copies in the RNA world , in Science , vol. 292, n. 5520, 2001, p. 1278, PMID 11360970 .
- ^ Szathmáry E, What is the optimum size for the genetic alphabet? ( PDF ), in Proc Natl Acad Sci USA , vol. 89, n. 7, 1992, pp. 2614–8, PMID 1372984 .
- ^ Lindahl T, Instability and decay of the primary structure of DNA , in Nature , vol. 362, n. 6422, 1993, pp. 709–15, PMID 8469282 .
- ^ Vreeland R, Rosenzweig W, Powers D, Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal , in Nature , vol. 407, n. 6806, 2000, pp. 897–900, PMID 11057666 .
- ^ Hebsgaard M, Phillips M, Willerslev E, Geologically ancient DNA: fact or artefact? , in Trends Microbiol , vol. 13, n. 5, 2005, pp. 212–20, PMID 15866038 .
- ^ Nickle D, Learn G, Rain M, Mullins J, Mittler J, Curiously modern DNA for a "250 million-year-old" bacterium , in J Mol Evol , vol. 54, n. 1, 2002, pp. 134–7, PMID 11734907 .
- ^ Goff SP, Berg P, Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells , in Cell , vol. 9, 4 PT 2, 1976, pp. 695–705, PMID 189942 .
- ^ Houdebine L, Transgenic animal models in biomedical research , in Methods Mol Biol , vol. 360, pp. 163–202, PMID 17172731 .
- ^ Daniell H, Dhingra A, Multigene engineering: dawn of an exciting new era in biotechnology , in Curr Opin Biotechnol , vol. 13, n. 2, 2002, pp. 136–41, PMID 11950565 .
- ^ Job D, Plant biotechnology in agriculture , in Biochimie , vol. 84, n. 11, 2002, pp. 1105–10, PMID 12595138 .
- ^ Collins A, Morton N, Likelihood ratios for DNA identification ( PDF ), in Proc Natl Acad Sci USA , vol. 91, n. 13, 1994, pp. 6007–11, PMID 8016106 .
- ^ Weir B, Triggs C, Starling L, Stowell L, Walsh K, Buckleton J, Interpreting DNA mixtures , in J Forensic Sci , vol. 42, n. 2, 1997, pp. 213–22, PMID 9068179 .
- ^ Jeffreys A, Wilson V, Thein S, Individual-specific 'fingerprints' of human DNA. , in Nature , vol. 316, n. 6023, pp. 76–9, PMID 2989708 .
- ^ Baldi, Pierre. Brunak, Soren. Bioinformatics: The Machine Learning Approach MIT Press (2001) ISBN 978-0-262-02506-5
- ^ Gusfield, Dan. Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology . Cambridge University Press, 15 January 1997. ISBN 978-0-521-58519-4 .
- ^ Sjölander K, Phylogenomic inference of protein molecular function: advances and challenges , in Bioinformatics , vol. 20, n. 2, 2004, pp. 170-9, PMID 14734307 .
- ^ location Mount DM, Bioinformatics: Sequence and Genome Analysis , 2ª ed., Cold Spring Harbor Laboratory Press, 2004, ISBN 0-87969-712-1 .
- ^ Immagine prelevata da Strong M. Protein nanomachines. PLoS Biol. 2004 Mar;2(3):E73. Epub 2004 Mar 16. Entrez PubMed 15024422
- ^ Adleman L, Molecular computation of solutions to combinatorial problems , in Science , vol. 266, n. 5187, 1994, pp. 1021–4, PMID 7973651 .
- ^ Parker J, Computing with DNA. , in EMBO Rep , vol. 4, n. 1, 2003, pp. 7–10, PMID 12524509 .
- ^ Ashish Gehani, Thomas LaBean and John Reif. DNA-Based Cryptography . Proceedings of the 5th DIMACS Workshop on DNA Based Computers, Cambridge, MA, USA, 14–15 June 1999.
- ^ Russel P. Goodman, Schaap, Iwan AT; Tardin, CF; Erben, Christof M.; Berry, Richard M.; Schmidt, CF; Turberfield, Andrew J., Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication , in Science , vol. 310, n. 5754, 9 dicembre 2005, pp. 1661–1665, Bibcode : 2005Sci...310.1661G , DOI : 10.1126/science.1120367 , PMID 16339440 .
- ^ T. Hodge and MJTV Cope (2000). "A Myosin Family Tree". Journal of Cell Science 113: 3353-3354 Entrez PubMed 10984423
- ^ Wray G, Dating branches on the tree of life using DNA , in Genome Biol , vol. 3, n. 1, 2002, pp. REVIEWS0001, PMID 11806830 .
- ^ I Letunic, Interactive Tree Of Life (iTOL): an online tool for phylogenetic tree display and annotation. , in Bioinformatics , 23(1), 2007, pp. 127-8.
- ^ FD Ciccarelli, Toward automatic reconstruction of a highly resolved tree of life. , in Science , 311(5765), 2006, pp. 1283-7.
- ^ Lost Tribes of Israel , NOVA, messa in onda sullaPBS : 22 febbraio 2000. Testo disponibile su PBS.org (ultimo accesso: 4 marzo 2006)
Bibliografia
- ( EN ) Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, Peter Walters, Molecular Biology of the Cell , New York e Londra, Garland Science, 2002, ISBN 0-8153-3218-1 .
- ( EN ) John M. Butler,Forensic DNA Typing , Elsevier, 2001, ISBN 978-0-12-147951-0 .
- ( EN ) Berg J., Tymoczko J., Stryer L., Biochemistry , WH Freeman and Company, 2002, ISBN 0-7167-4955-6 .
- ( EN ) Baldi, Pierre. Brunak, Soren, Bioinformatics: The Machine Learning Approach , MIT Press, 2001, ISBN 978-0-262-02506-5 .
- Gusfield, Dan, Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology , Cambridge University Press, 15 gennaio 1997, ISBN 978-0-521-58519-4 .
- ( EN ) location Mount DM, Bioinformatics: Sequence and Genome Analysis , 2ª ed., Cold Spring Harbor Laboratory Press, 2004, ISBN 0-87969-712-1 .
- ( EN ) Andrew Berry; James D. Watson , DNA: the secret of life , New York, Alfred A. Knopf, 2003, ISBN 0-375-41546-7 .
- ( EN ) James D. Watson , DNA: The Secret of Life , Random House, 2004, ISBN 978-0-09-945184-6 .
- ( EN ) Robert Olby, Francis Crick: A Biography , Plainview, NY, Cold Spring Harbor Laboratory Press, 2009, ISBN 0-87969-798-9 .
- ( EN ) Gunther Stent ; James D. Watson , The Double Helix , New York, Norton, 1980, ISBN 0-393-95075-1 .
- John W. Pelley, Biochimica , Elsevier, 2008, ISBN 978-88-214-3020-6 .
- Giuliano Ricciotti, Biochimica di base , Zanichelli, 1987, ISBN 88-08-01182-8 .
Voci correlate
- Acido nucleico
- Complementarità
- Computer a DNA
- Conformazioni del DNA
- Cromosoma
- DNA a tripla elica
- DNA clamp
- DNA complementare
- DNA girasi
- DNA metiltransferasi
- DNA mitocondriale
- DNA non codificante
- DNA nucleotidil-esotrasferasi
- DNA polimerasi (DNA-dipendente)
- DNA polimerasi (RNA-dipendente)
- DNA ricombinante in botanica
- DNA ricombinante
- DNA satellite
- DNA-binding protein
- Dogma centrale della biologia molecolare
- Frammentazione del DNA
- Impronta genetica
- Macchina di DNA
- Nanotecnologia del DNA
- Prodotto genico
- Progetto genoma
- RNA
- Sequenza ripetuta di DNA
- Superavvolgimento del DNA
- Tecnologia del DNA ricombinante
- Traduzione (biologia)
- Trascrizione (biologia)
- Virus a DNA
- DNA Nucleare
Altri progetti
-
 Wikiquote contiene citazioni di o su DNA
Wikiquote contiene citazioni di o su DNA -
 Wikibooks contiene testi o manuali su DNA
Wikibooks contiene testi o manuali su DNA -
 Wikizionario contiene il lemma di dizionario « DNA »
Wikizionario contiene il lemma di dizionario « DNA » -
 Wikimedia Commons contiene immagini o altri file su DNA
Wikimedia Commons contiene immagini o altri file su DNA - Wikibooks - Analisi del proprio genoma
Collegamenti esterni
- DNA , su Treccani.it – Enciclopedie on line , Istituto dell'Enciclopedia Italiana .
- ( EN ) DNA , su Enciclopedia Britannica , Encyclopædia Britannica, Inc.
- ( EN ) DNA Interactive , su dnai.org .
| Controllo di autorità | Thesaurus BNCF 5481 · LCCN ( EN ) sh85037008 · GND ( DE ) 4070512-2 · BNF ( FR ) cb119649178 (data) · BNE ( ES ) XX527602 (data) · NDL ( EN , JA ) 00561484 |
|---|